DRUGONE
细胞的功能角色由其分子身份和空间环境的复杂交互所决定,但在现有的空间建模中,这种关系往往被掩盖。为在计算机中高效处理大规模异质性空间数据,研究人员提出了 DECIPHER,该方法通过一种新颖的跨尺度对比学习策略,将细胞的细胞内和细胞外表征进行解缠表示。系统性基准测试与多种真实案例研究表明,DECIPHER 生成的解缠嵌入能够清晰描绘跨尺度的细胞—环境互作。值得注意的是,DECIPHER 具有高度可扩展性,可处理包含数百万细胞的空间图谱,这在现有方法中基本不可行。

在多细胞生物中,细胞必须通过相互作用组织形成三维组织结构。单个细胞的空间环境与其固有属性同等重要,共同决定其生理功能及潜在病理变化。空间组学技术的兴起使得研究人员能够同时刻画分子身份和空间背景,但如何在计算机中准确表征这两方面仍然是重大挑战。
常用策略是 嵌入学习,即将高维空间数据映射为低维向量,从而增强生物学信号并减少技术噪声。受单细胞组学成功经验的启发,现有空间分析方法通常尝试将转录组和空间信息共同编码为一个整体嵌入,用于后续聚类、批次校正或切片比对。然而,这种整体嵌入方式不可避免地将分子身份与空间环境纠缠在一起,难以直接剖析两者关系,也与研究细胞通信的需求存在脱节。同时,大多数基于图神经网络的方法在扩展至百万级细胞数据时面临严重的计算瓶颈。
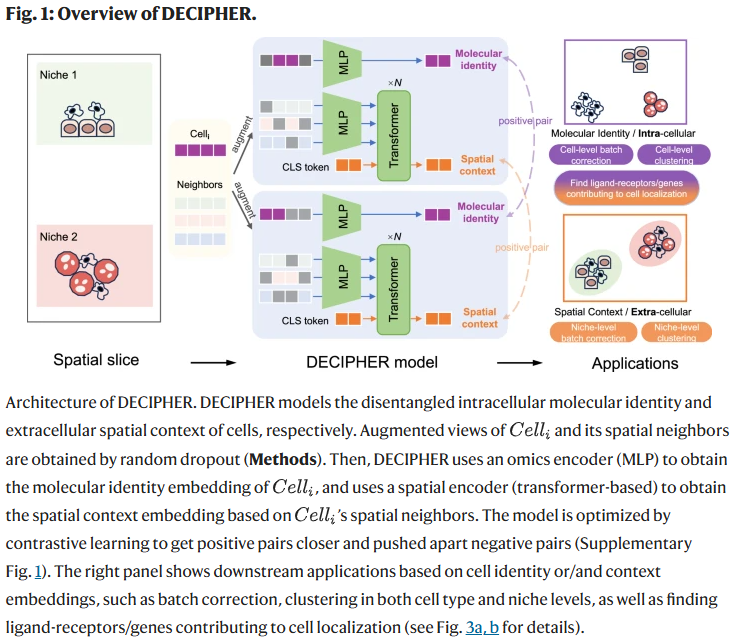
针对这些问题,研究人员开发了 DECIPHER ——一种面向大规模异质空间数据的上下文感知深度学习模型。DECIPHER 采用跨尺度对比学习框架,分别学习细胞的细胞内(分子身份)与细胞外(空间环境)嵌入,且基于 Transformer 架构实现了显著的扩展能力。除了在基准评测中表现优越,DECIPHER 还可用于实际生物学问题,例如揭示 B 细胞在生发中心的定位机制,以及识别肿瘤微环境中免疫细胞浸润的关键基因。
结果
解缠式细胞内与细胞外嵌入
与追求单一整体嵌入的传统方法不同,DECIPHER 由两个互相关联的模块构成:组学编码器(提取分子身份特征)与 空间编码器(捕捉邻近细胞的空间信息)。两者通过跨尺度对比学习策略协同优化,从而在保持共享信息的同时保留特定差异。训练完成后,所得嵌入分别适用于细胞类型识别或空间领域划分等任务。
基准评测中的优越表现
研究人员将 DECIPHER 与多种先进方法对比,涵盖空间建模、单细胞整合及批次校正等场景。在多种真实与模拟数据集中,DECIPHER 的空间嵌入在聚类一致性指标上均优于其他方法,尤其在百万级细胞数据上保持了稳定性能。相比之下,许多图神经网络方法在大规模数据上因内存溢出或运行时间过长而失败。
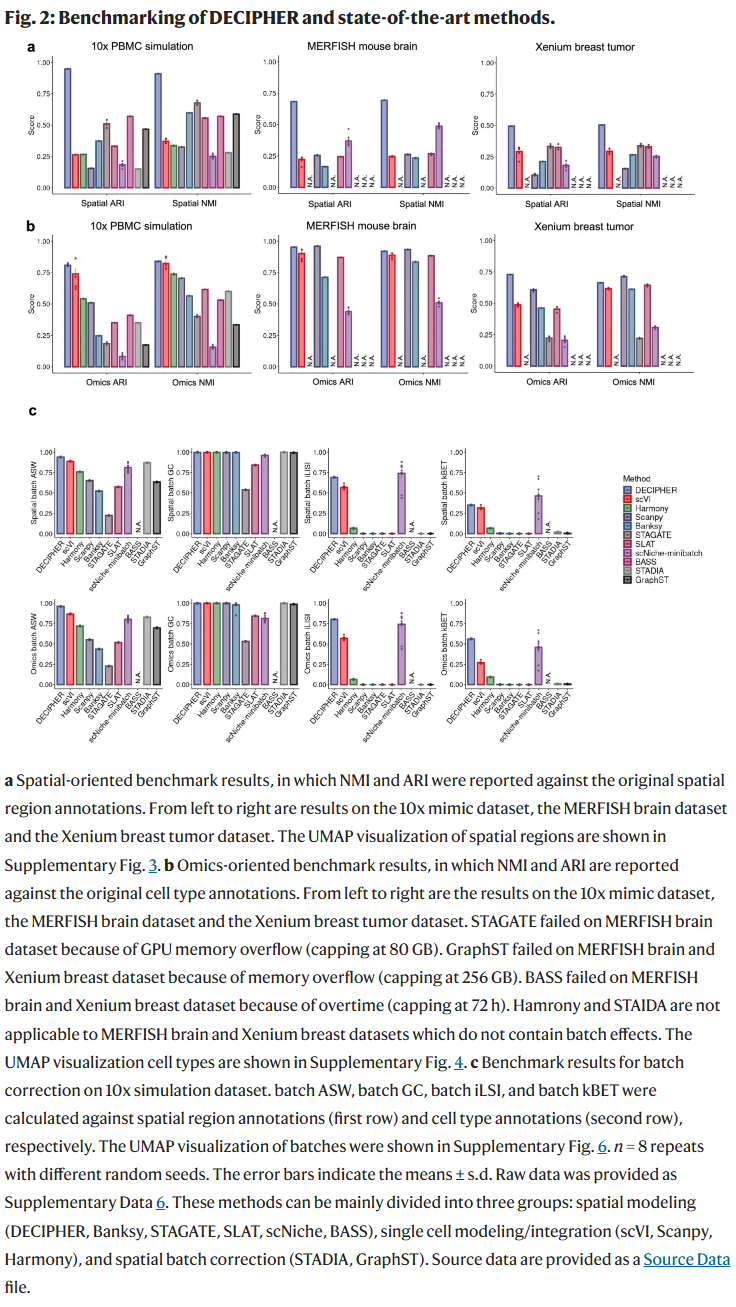
此外,在组学导向的指标上,DECIPHER 同样优于整体嵌入方法,表明其解缠设计能够更好地区分分子和空间两方面的特征。其批次校正效果也显著优于现有空间方法,并与单细胞专用的先进工具相当。
揭示跨尺度的细胞—环境互作
DECIPHER 的解缠嵌入使研究人员能够直接建模复杂的细胞—环境互作。以淋巴结数据为例,DECIPHER 准确识别出 B 细胞定位过程中关键的配体—受体对(如 CXCL12_CXCR4 与 CXCL13_CXCR5),而这些信号常被传统方法遗漏。在乳腺癌空间数据中,DECIPHER 发现 PTGDS 等基因与免疫细胞浸润强相关,进一步验证了其在有限基因覆盖场景下的独特优势。
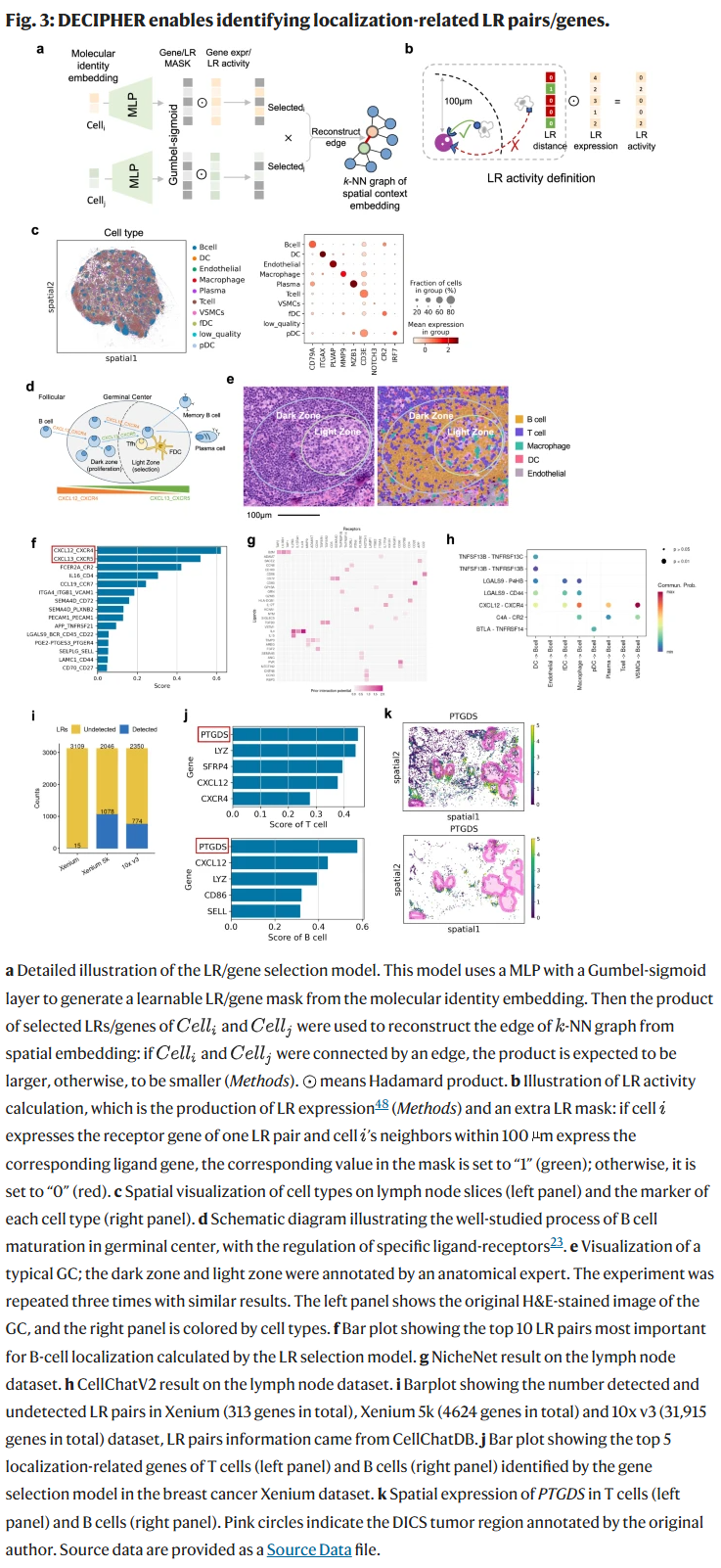
空间图谱级别分析
在 人类泛癌空间图谱(870 万细胞,八种癌症类型)中,DECIPHER 成功校正批次效应,并解析出肿瘤核心、低淋巴浸润与高淋巴浸润三类生态位,揭示了免疫细胞空间分布与功能状态的耦合关系。运行效率方面,DECIPHER 在单卡 GPU 上即可在 4 小时内完成分析,远超现有方法的可扩展性。
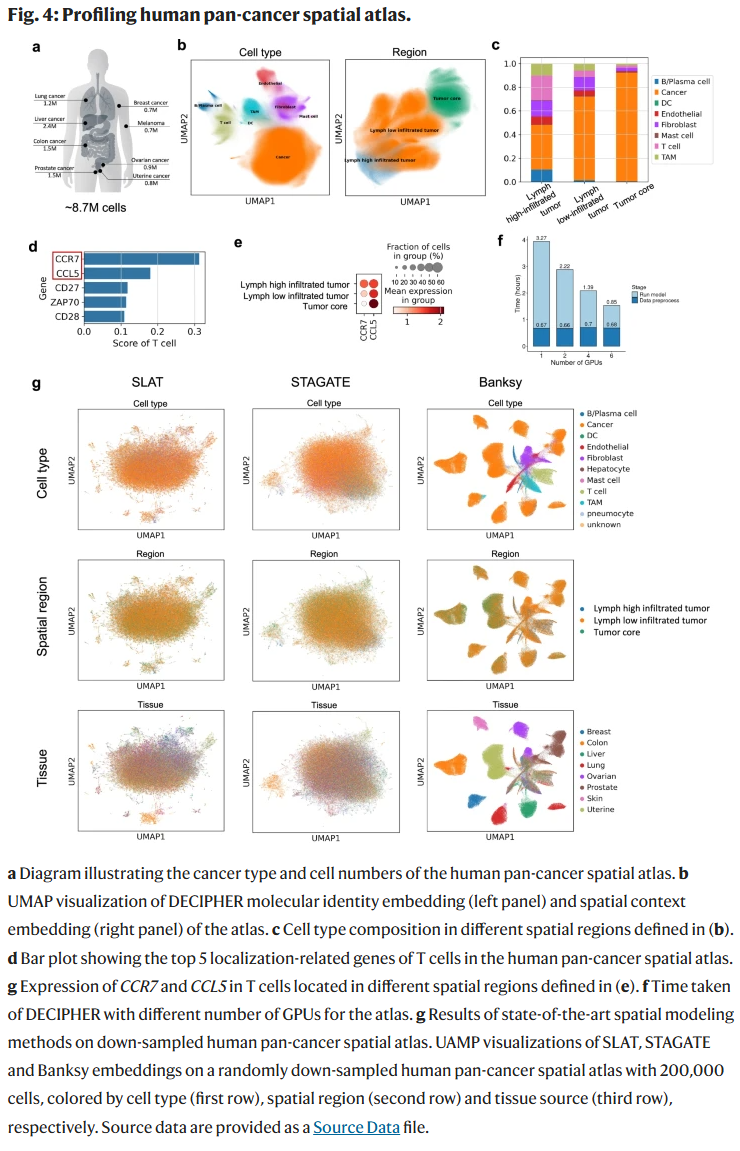
在 小鼠大脑三维图谱(350 万细胞,151 连续切片)中,DECIPHER 的三维嵌入能够准确还原脑区解剖结构,而仅基于二维切片信息的分析无法分辨主要脑区。这凸显了三维建模在解析复杂组织空间格局中的重要性。
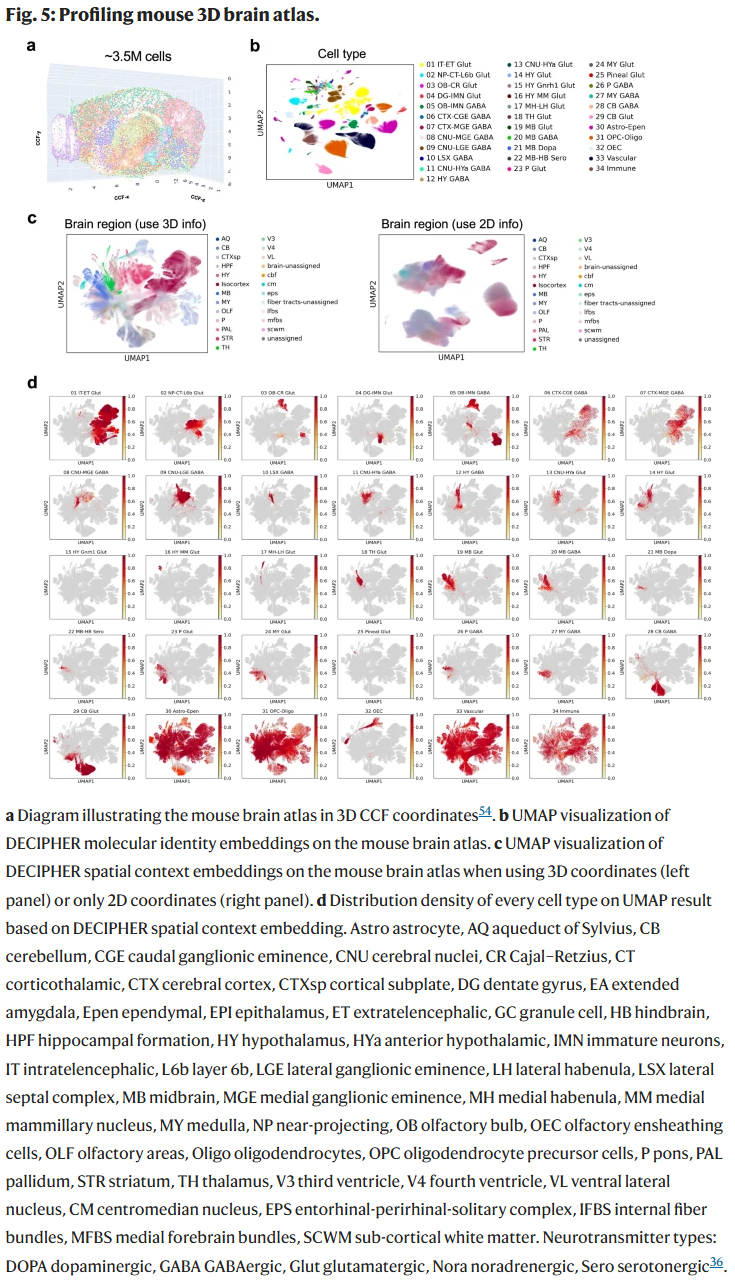
讨论
细胞的内在分子身份与外部空间环境密切关联,但不应被混为一体。研究人员提出的 DECIPHER 通过跨尺度对比学习框架,实现了高保真、解缠的双重嵌入。其优势不仅体现在基准和案例中的优越表现,更在于能够量化与空间定位高度相关的分子因子,从而深化对细胞调控机制的理解。
随着空间组学技术的通量持续提升,扩展性成为方法学发展的关键挑战。DECIPHER 采用轻量化的 Transformer 空间编码器和低维嵌入设计,兼顾性能与计算效率,并可通过多 GPU 并行进一步加速。其自监督学习策略和架构特征使其具备成为未来大规模空间组学预训练模型基础的潜力。
尽管如此,DECIPHER 仍有局限。例如,当前仅采用随机 dropout 作为数据增强,未来可探索更丰富的策略;基准测试仅涵盖 Xenium 与 MERFISH 技术,尚需更多高质量空间数据进行全面评估。
总体而言,DECIPHER 是一款高性能、高可扩展性的空间组学分析工具,为探索细胞在原生组织环境中的功能提供了新的视角,并为未来构建空间组学基础模型奠定了方法学基础。
整理 | DrugOne团队
参考资料
Xia, CR., Cao, ZJ. & Gao, G. DECIPHER for learning disentangled cellular embeddings in large-scale heterogeneous spatial omics data. Nat Commun 16, 7991 (2025).
https://doi.org/10.1038/s41467-025-63140-8

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢