DRUGAI
靶向蛋白降解(Targeted Protein Degradation, TPD)是当前药物研发的前沿方向,其中分子胶降解剂(Molecular Glue Degraders, MGDs)与蛋白降解靶向嵌合体(Proteolysis-Targeting Chimeras, PROTACs)是最具代表性的两类技术。两者均通过泛素-蛋白酶体系统实现靶蛋白的选择性降解,但机制差异显著:PROTAC借助双功能连接子桥接E3连接酶与靶蛋白,而MGD以小巧的单价分子诱导三元复合物形成,具备更优的药物样性质。近年来已有20余个分子胶进入临床试验,展现出巨大应用潜力。
然而,分子胶降解剂的发现与理性设计面临巨大挑战。与相对模块化的PROTAC(蛋白降解靶向嵌合体)不同,分子胶作用机制依赖复杂的蛋白-蛋白相互作用,其发现往往依赖偶然发现或大规模筛选。如何系统整合研究数据,为分子胶的理性设计提供支撑,是该领域亟待解决的问题。
2025年8月5日,中国海洋大学医药学院博士后王霄与硕士研究生庄智尧作为本研究共同第一作者,秦冲教授、房森彪和徐锡明研究员作为共同通讯,在Nucleic Acids Research上发表了题为“MolGlueDB: An Online Database of Molecular Glues”的研究论文(DOI: 10.1093/nar/gkaf811)。该工作构建了首个专注于MGDs的在线数据库MolGlueDB,整合了2001年1月至2025年5月期间241篇文献,包含1840个条目、1629个独特MGDs、28个招募蛋白和94个靶点。数据库涵盖化学结构、结合亲和力、降解能力、生物活性、理化性质等,支持文本/结构搜索,可极大促进MGDs的理性设计与药物发现。


本研究针对分子胶降解剂(MGDs)数据碎片化与整合难题,创新构建了MolGlueDB数据库。该数据库全面涵盖经典单价MGDs(如沙利度胺诱导CRBN降解IKZF1/3)、双价非共价/共价MGDs(如IBG3招募DCAF16降解BRD4)以及表现出MGD-like行为的PROTACs(如GBD-9双重降解BTK和GSPT1)。此外,数据库还纳入无降解活性的分子胶候选物,提供结构-活性关系(SAR)关键洞见,并具备扩展潜力,未来可涵盖非降解分子胶,进一步拓宽应用边界。
数据库构建过程分为三个关键步骤:①数据收集阶段,通过关键词检索PubMed等平台,筛选241篇文献(涵盖79种期刊),构建高质量数据集;②数据处理阶段,提取核心信息包括SMILES化学结构、DC50/Dmax降解参数、IC50/Kd结合亲和力、靶点与招募蛋白、PDB晶体结构、PK/PD药代数据以及蛋白质组学结果,并利用RDKit工具计算分子量、LogP等理化性质;③数据库构建阶段,采用Vue 3前端框架、Flask后端服务和MySQL存储系统,支持Ketcher分子编辑器,实现高效数据管理和交互。
MolGlueDB的核心特征包括:灵活的文本与结构搜索(支持子结构和相似性查询)、高级过滤功能(可按分子量范围、降解活性等多维度筛选)、详尽的条目页面(展示分子结构、属性参数、相关文献及PDB复合物如CRBN-IMiD-IKZF1三元结构),以及SDF/CSV格式的数据导出,便于用户深度分析与应用。
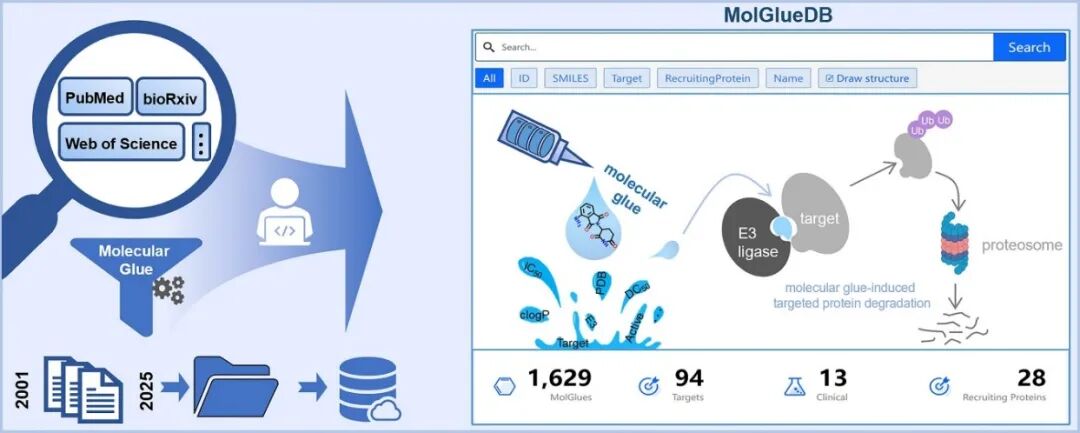
图1 MolGlueDB的整体架构与设计理念
MolGlueDB的主要亮点包括:
(1) 数据规模宏大:整合了2001年1月至2025年5月的241篇文献,收录了1840条信息,涵盖了1629种独特分子胶降解剂(MGDs)、28种E3招募蛋白和94个靶点,构建了全面的数据资源。
(2) 信息维度丰富:包含化学结构、结合亲和力、降解能力、生物活性及理化性质等多维度数据,并持续扩展药代动力学(PK/PD)相关信息,为深入研究提供坚实基础。
(3) 功能模块强大:支持文本与结构双重搜索,提供子结构查询、分子编辑器及多维度筛选功能(包括靶点、E3连接酶、理化性质、药效团类型等),极大提升用户体验与分析效率。
(4) 动态优化升级:MolGlueDB于2025年初上线并开源且持续更新完善,并后期将增强对人工智能驱动的分子胶理性设计的支持能力,确保数据与技术的前沿性。
在实证分析中,MolGlueDB整合了临床MGDs(如靶向血液瘤的IMiDs衍生物)及新兴MGDs(如靶向KRAS的固瘤分子胶),为三元复合物预测模型提供了坚实的数据支撑。相较于传统工具,MolGlueDB显著提升了MGD发现效率与机制研究的深度。为加速科研转化,研究团队已通过https://www.molgluedb.com开源数据库,确保全球研究者可自由访问并参与数据更新,助力靶向蛋白降解领域的持续创新。
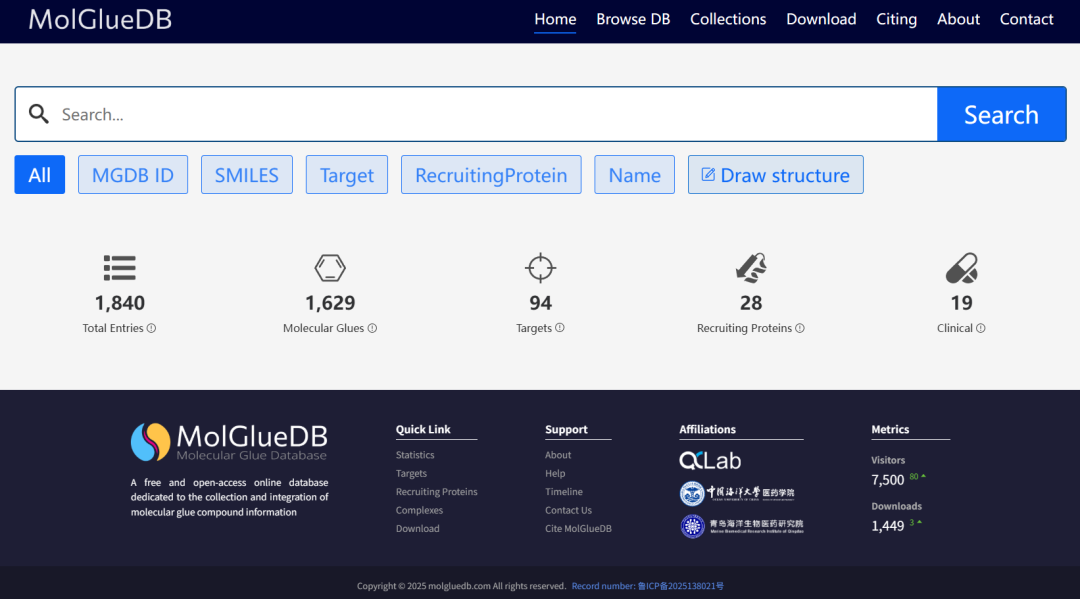
图 2 MolGlueDB在线数据库的主页界面
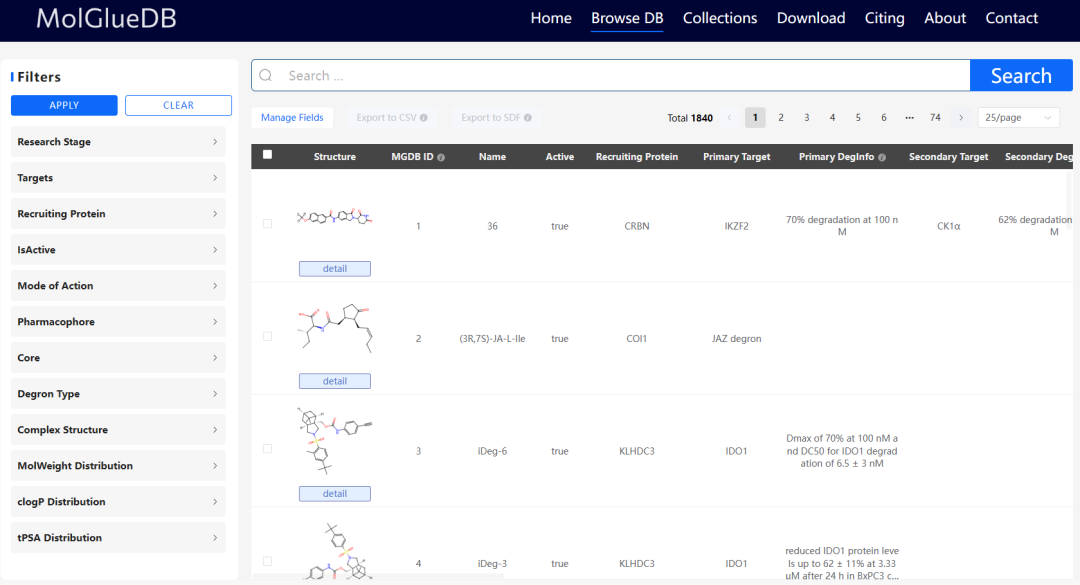
图 3 MolGlueDB数据库页面
自2025年8月部署以来,MolGlueDB 访问量已累计超过 7500 次,数据下载与导出次数达 1400 余次,充分表明该平台已在学术界和产业界获得积极关注与广泛应用。作为全球首个分子胶数据库,MolGlueDB 将为靶向蛋白降解研究提供坚实的数据基础和便捷的工具平台,加速分子胶药物的理性设计与新药研发。
本研究的共同第一作者为中国海洋大学医药学院博士后王霄与硕士研究生庄智尧,论文共同通讯作者为徐锡明、房森彪和秦冲教授。该工作得到山东省重点研发计划 [项目号:2022CXPT038] 以及青岛市科技示范工程项目 [项目号:25-1-1-sfgc-2-gx] 的资助。
文章结论与讨论,(未来)启发与展望
该研究开发了MolGlueDB,全球首个人工分子胶降解剂(MGD)专用数据库,整合多维度数据,支持高级搜索功能,为理性药物设计提供强有力工具。该工作不仅彰显了MGDs在临床应用的显著进展与独特优势,还揭示了CRBN主导的现状及其多靶点特性,为拓展E3连接酶库和深入机制研究提供了关键的结构-活性关系(SAR)洞见。然而,数据库当前存在一定局限性:主要聚焦于降解型MGDs,尚未纳入非降解分子胶;数据来源依赖已发表文献,可能遗漏未公开的新兴MGDs;未整合人工智能预测工具;临床及药代动力学(PK)数据覆盖尚不全面。这些局限性引出了新的科学问题:如何实现数据的动态更新?如何扩展至非降解分子胶及更广泛的E3连接酶?展望未来,可通过社区协作实现数据实时更新,融入机器学习算法以预测三元复合物稳定性,并扩展数据库覆盖广义分子胶(如靶向KRAS的RMC-6291)。这些改进将推动靶向蛋白降解(TPD)领域向多模态药物研发转型,加速从偶然发现向理性设计的转变,为癌症、神经退行性疾病等重大疾病的治疗带来新的希望。
撰文︱房森彪
审阅︱秦 冲
参考资料
Xiao Wang, Zhiyao Zhuang, Chengwei Zhang, Bowen Zhang, Wei Zhan, Yifan Wang, Zhaojuan Liu, Shanwen Yuan, Wenjia Niu, Qi He, Yanqing Tian, Ximing Xu, Senbiao Fang, Chong Qin, MolGlueDB: an online database of molecular glues, Nucleic Acids Research, 2025;, gkaf811.
https://doi.org/10.1093/nar/gkaf811
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢