
导语
在科学研究迈向智能化的当下,大语言模型正逐步打破传统AI仅擅长文本和图像生成的固有边界,深入渗透到材料科学、生物医药与复杂系统预测的核心领域。本期图智决策精选的Nature系列前沿研究,这些工作展现了“语言即物质结构”、“文本即生物功能”的跨模态生成范式:CrystaLLM将晶体结构建模转化为自回归文本生成问题,成功合成出多种未见于训练集的稳定新材料;ProteinDT引入多模态预训练框架,首次实现由自然语言引导的蛋白质设计;Token-Mol打通分子3D构象生成与属性预测,用token统一编码结构与性质,在速度与性能上全面超越扩散模型;TrinityLLM借助物理合成数据,实现对数据稀缺下聚合物性能的精确预测。
与此同时,科学大语言模型也在向“可解释科学发现”迈进:LLM4SD挖掘文献与分子数据的符号逻辑,用规则驱动分子性质建模;PandemicLLM则将疫情预测重构为文本推理任务,融合政策、行为、基因等非结构信息实现高精度预测;SciToolAgent则基于知识图谱自动编排数百种科学工具,重塑多工具科学工作流的交互范式。这些研究共同揭示了一个趋势:大语言模型不仅能“说出科学”,更正在“生成科学”与“发现科学”,AI for Science 正迎来从“跨模态表征”到“跨学科推理”的系统跃迁。
本文聚焦于大语言模型赋能的科学发现,遴选了7篇来自Nature系期刊中关于这一话题的文章。
研究领域:大语言模型,分子网络,蛋白质合成,多智能体,疫情预测

曾利丨编辑
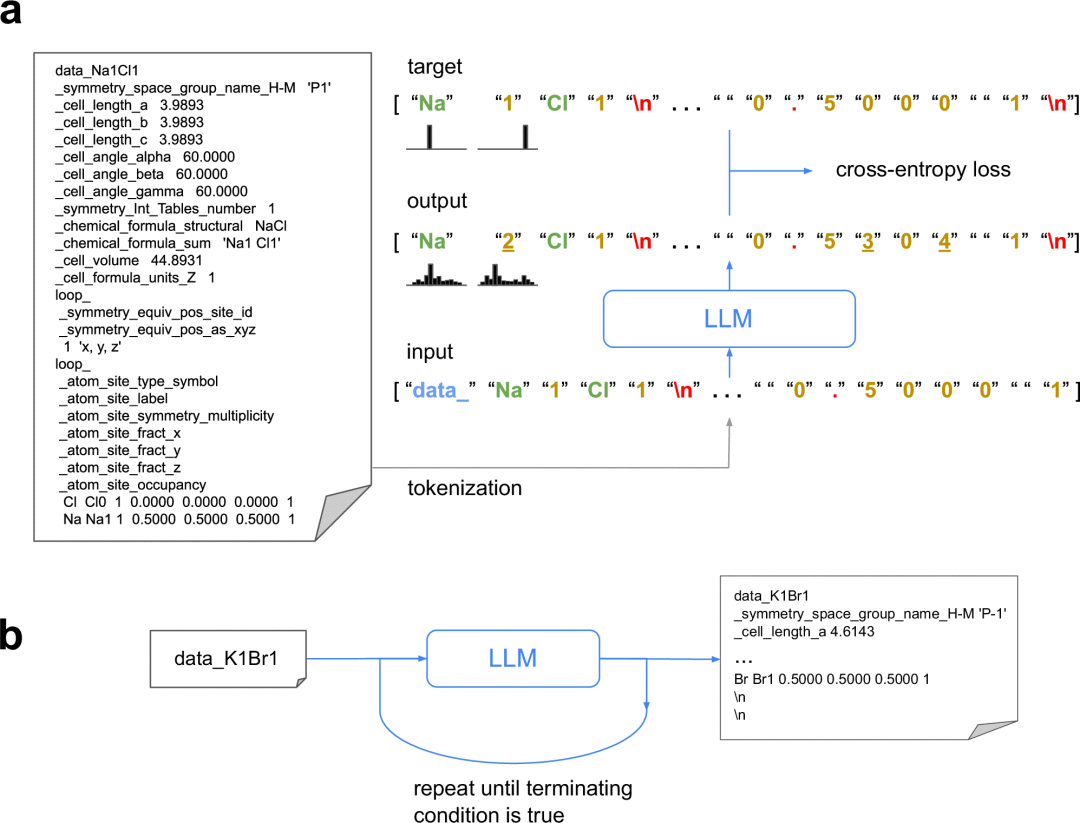
论文一:基于自回归大模型的晶体结构生成
论文一:基于自回归大模型的晶体结构生成
论文题目:Crystal structure generation with autoregressive large language modeling
基于自回归大模型的晶体结构生成
论文来源:Nature Communications
论文链接:https://www.nature.com/articles/s41467-024-54639-7
代码链接:https://github.com/lantunes/CrystaLLM
一作单位:University of Reading,UK

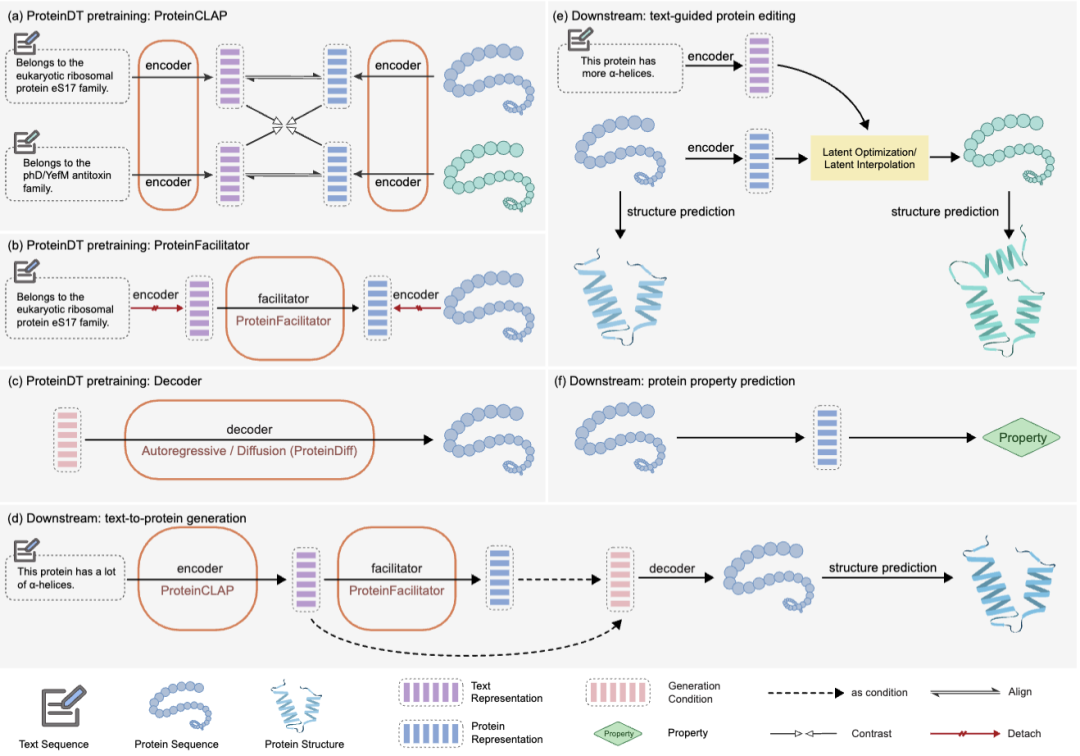
论文二:基于文本数据进行蛋白质设计的多模态框架
论文二:基于文本数据进行蛋白质设计的多模态框架
论文题目:A text-guided protein design framework
基于文本数据进行蛋白质设计的多模态框架
论文来源:Nature Machine Intelligence
论文链接:https://www.nature.com/articles/s42256-025-01011-z
代码链接:https://github.com/chao1224/ProteinDT
一作单位:University of California Berkeley, USA

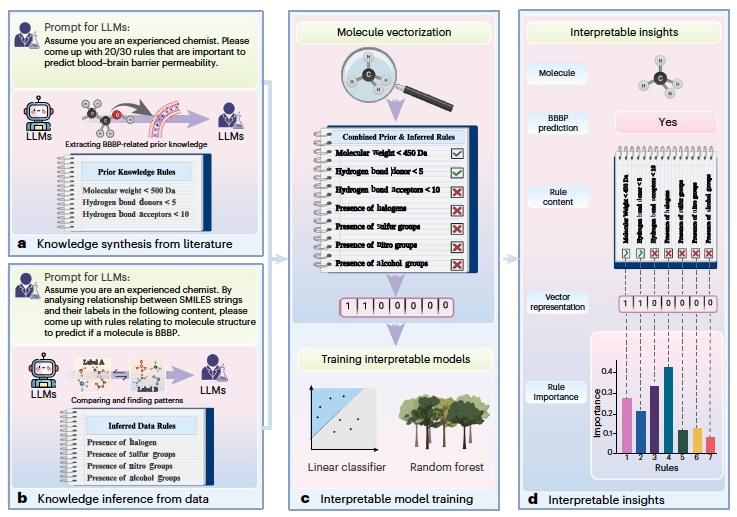
论文三:用于分子性质预测的科学发现大模型
论文三:用于分子性质预测的科学发现大模型
论文题目:Large language models for scientific discovery in molecular property prediction
用于分子性质预测的科学发现大模型
论文来源:Nature Machine Intelligence
论文链接:https://www.nature.com/articles/s42256-025-00994-z
代码链接:https://github.com/zyzisastudyreallyhardguy/LLM4SD
一作单位:Monash University,Australia

论文四:知识图谱驱动的多工具集成科学智能体
论文四:知识图谱驱动的多工具集成科学智能体
论文题目:SciToolAgent: a knowledge-graph-driven scientific agent for multitool integration
知识图谱驱动的多工具集成科学智能体
论文来源:Nature Computational Science
论文链接:https://www.nature.com/articles/s43588-025-00849-y
代码链接:https://github.com/hicai-zju/scitoolagent
一作单位:Zhejiang University, China

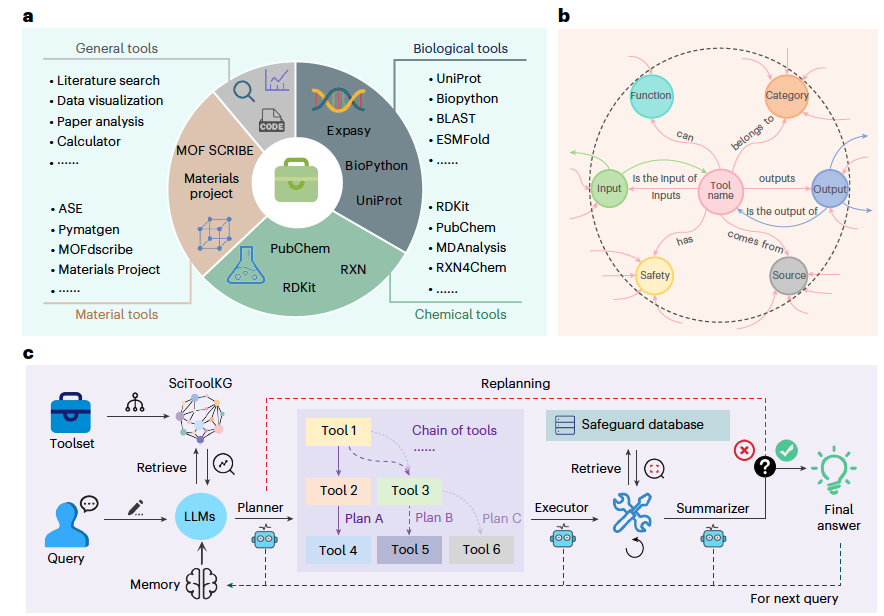
核心图:SciToolAgent 概览。(a) 研究中使用的工具集,其中包括通用工具以及常用的生物学、化学和材料相关工具。(b) SciToolKG 的模式架构,涵盖了每个工具的多样信息,如输入/输出格式、特定功能、安全等级等。(c) SciToolAgent 的整体工作流程:在接收到用户查询后,基于 LLM 的 Planner(规划器) 会利用基于 SciToolKG 的检索增强生成(RAG)来生成工具链,随后由基于 LLM 的 Executor(执行器) 顺序执行。接着通过 安全检查模块 调用安全数据库,以确保生成的解决方案符合伦理与安全要求。最后,基于 LLM 的 Summarizer(总结器) 对执行结果进行总结,评估问题求解过程,并在必要时提示 Planner 生成新的工具链。最终答案会被存储到 记忆模块 中,作为下一次查询的上下文。(arxiv版的图,对比了,没什么出入)
论文五:基于大语言模型的分子化药物设计
论文五:基于大语言模型的分子化药物设计
论文题目:Token-Mol 1.0: tokenized drug design with large language models
基于大语言模型的分子化药物设计
论文来源:Nature Communications
论文链接:https://www.nature.com/articles/s41467-025-59628-y
代码链接:https://github.com/hicai-zju/scitoolagent
一作单位:Zhejiang University, China

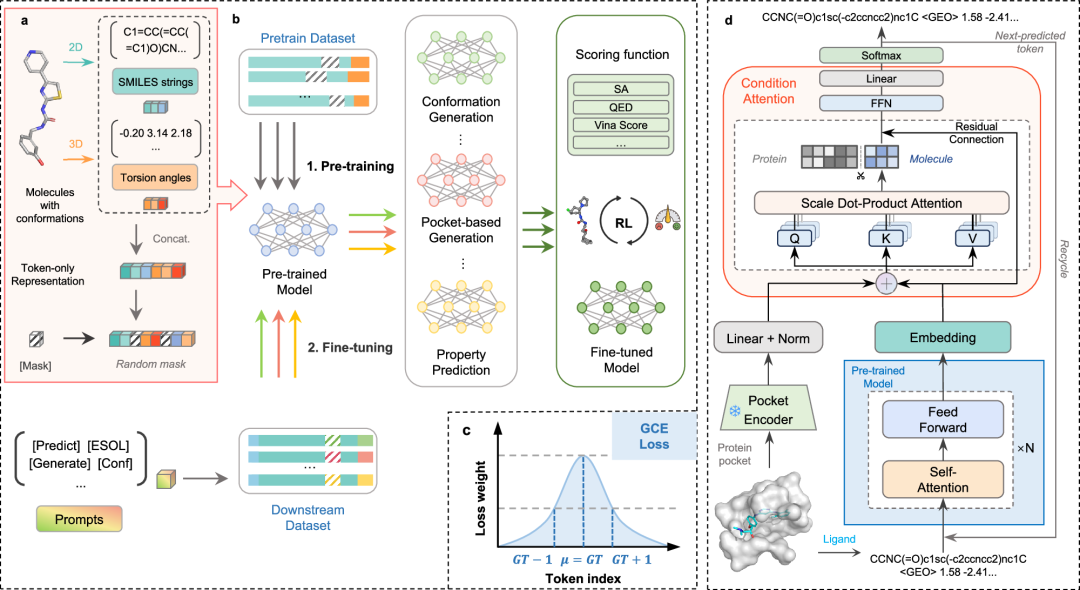

论文六:数据稀缺条件下基于大模型的聚合物性质学习
论文六:数据稀缺条件下基于大模型的聚合物性质学习
论文题目:Harnessing large language models for data-scarce learning of polymer properties
数据稀缺条件下基于大模型的聚合物性质学习
论文来源:Nature Computational Science
论文链接:https://www.nature.com/articles/s43588-025-00768-y
代码链接:https://github.com/ningliu-iga/TrinityLLM
一作单位:Global Engineering and Materials Inc., USA

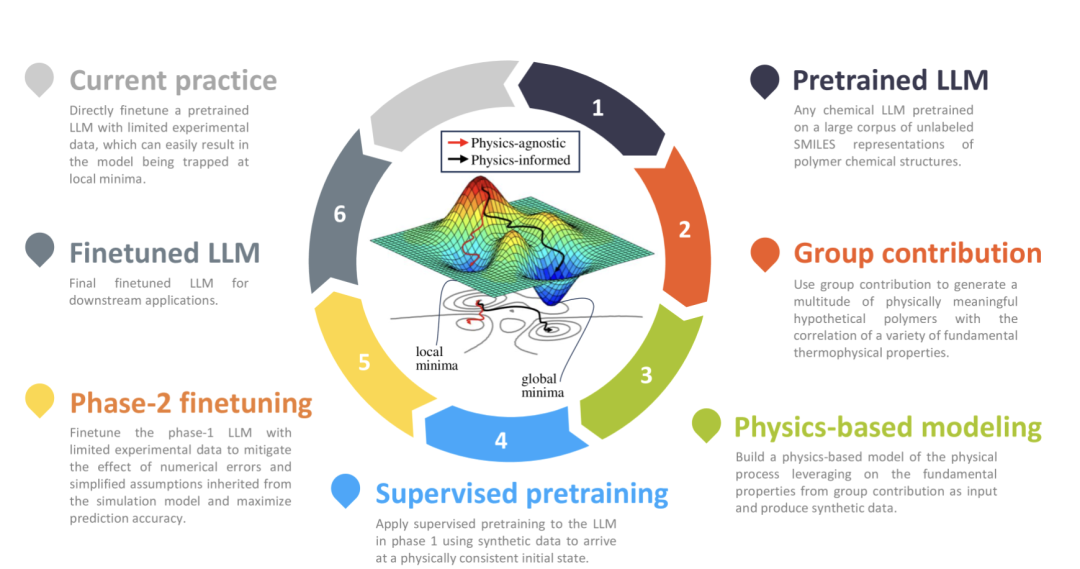
论文七:基于大语言模型推动传染病实时预测研究发展
论文七:基于大语言模型推动传染病实时预测研究发展
论文题目:Advancing real-time infectious disease forecasting using large language models
基于大语言模型推动传染病实时预测研究发展
论文来源:Nature Computational Science
论文链接:https://www.nature.com/articles/s43588-025-00798-6
代码链接:https://github.com/miemieyanga/PandemicLLM
一作单位:Johns Hopkins University, USA

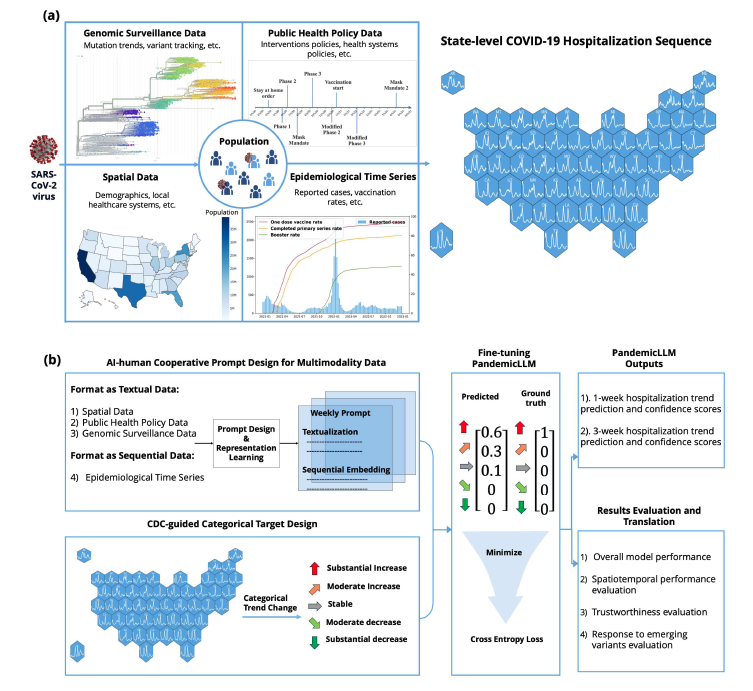
核心图1:PandemicLLMs 的疫情数据流与处理流程概览。(a) 多模态数据洞察(Multi-modality data insights):我们的多模态数据集整合了四类疫情相关数据源:空间数据(人口统计学与医疗保健指标)、流行病学时间序列(病例报告、住院人数和疫苗接种率)、公共卫生政策(以文本形式记录的政府干预措施)以及基因组监测数据(结合了变异株的文本描述与其流行情况的周度序列)。整个数据集包含 5200 条记录,覆盖美国 50 个州,时间跨度为 104 周。SARS-CoV-2 的系统发育树是使用 Nextstrain 生成的。(b) PandemicLLMs 的构建流程:为了预测疫情住院趋势,我们将问题形式化为一个序数分类任务。根据 CDC 指南,我们定义了五个类别:大幅下降、中度下降、稳定、中度上升、大幅上升。通过 AI–人类协作的提示设计将多模态数据转换为文本格式,PandemicLLMs 在这些提示与目标标签的基础上进行微调,以实现 1 周和 3 周的预测。我们特别强调对模型进行严格的性能评估,以验证预测的 准确性与可信度。
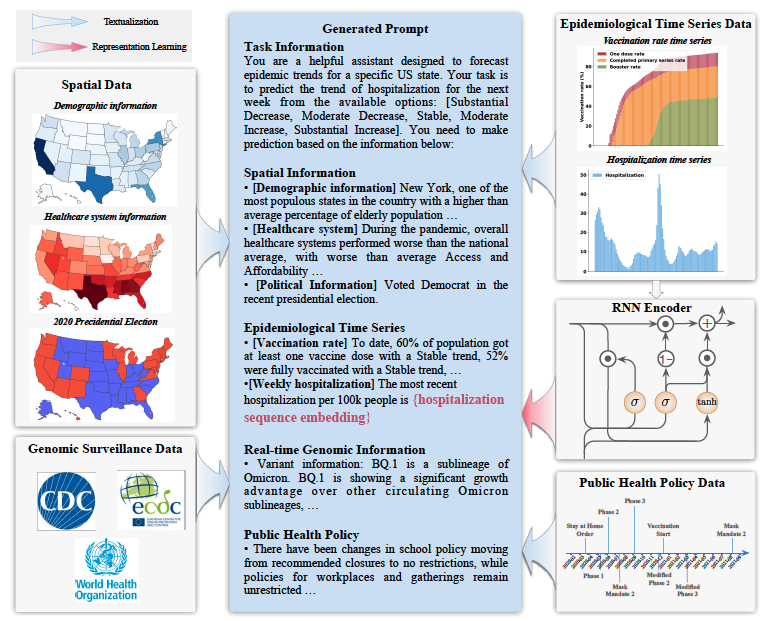
核心图2:AI–人类协作提示设计示意图。美国 50 个州的空间数据被转化为描述性文本,以反映其排名;政策数据包含防控强度及其逐周变化;流行病学时间序列数据结合了叙事生成与表征学习;基因组监测数据则将变异株特征的文本摘要与其最新流行情况结合。蓝色箭头表示信息文本化,红色箭头表示序列表征学习。每个设计的提示长度在 296 至 322 个词之间。
大模型与生物医学:
AI + Science第二季读书会

详情请见:
7. 集智学园精品课程免费开放,解锁系统科学与 AI 新世界
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢