DRUGONE
蛋白质工程在设计具有特定功能的蛋白质方面展现了巨大潜力,但由于可能的突变空间极为庞大,而实验室验证能力有限,往往难以高效发现最佳序列。为此,研究人员提出了 μProtein 框架,通过结合深度学习模型 μFormer(用于精确预测突变效应)与强化学习算法 μSearch(用于高效探索适应度景观),加速蛋白质工程进程。μProtein 能利用单点突变数据,预测并优化含有多位点突变的复杂序列,其关键在于对表观遗传交互作用(epistasis)的建模和多步搜索策略。在基准数据集上的表现验证了其有效性,并且在实验室中成功发现了具有显著功能增益的多点 β-内酰胺酶突变体,其活性超过已知最高水平。结果表明,μProtein 能够在广阔的蛋白质序列空间中发现具有潜在价值的突变,提供了一种稳健且高效的优化手段。

蛋白质工程作为生物技术的核心领域,旨在通过优化序列来获得定制化功能,推动药物研发、农业和新型生物酶的开发。核心挑战在于如何准确地将蛋白质序列映射到功能,即构建所谓的 适应度景观。这一景观往往复杂且崎岖,因为氨基酸残基之间存在非线性相互作用。
高通量实验方法如 深度突变扫描(DMS)和多重变体效应检测推动了单点突变效应的系统性研究。然而,它们仍存在局限:一是难以覆盖指数级增长的多重突变组合空间,二是依赖特定的实验读出系统(如细胞生长、荧光或结合亲和力),无法适用于所有功能表型。因此,基于有限实验数据的计算预测方法成为重要替代途径,能够探索超出现有实验范围的序列–功能关系。
μProtein 框架正是为此设计:μFormer 捕捉复杂的表观遗传效应,μSearch 则通过强化学习高效搜索序列空间。二者结合,为蛋白质工程提供了全面而高效的新策略。
结果
μProtein 总览
μProtein 通过 μSearch 与 μFormer 的协作,能够从有限实验数据中泛化,并探索广阔的适应度景观。μSearch 将蛋白质工程任务建模为马尔可夫决策过程,通过多步策略性突变,生成潜在序列;μFormer 则作为“预测先知”,为这些序列提供适应度评分。该框架能高效识别高适应度突变组合,并在实际实验中得到验证。
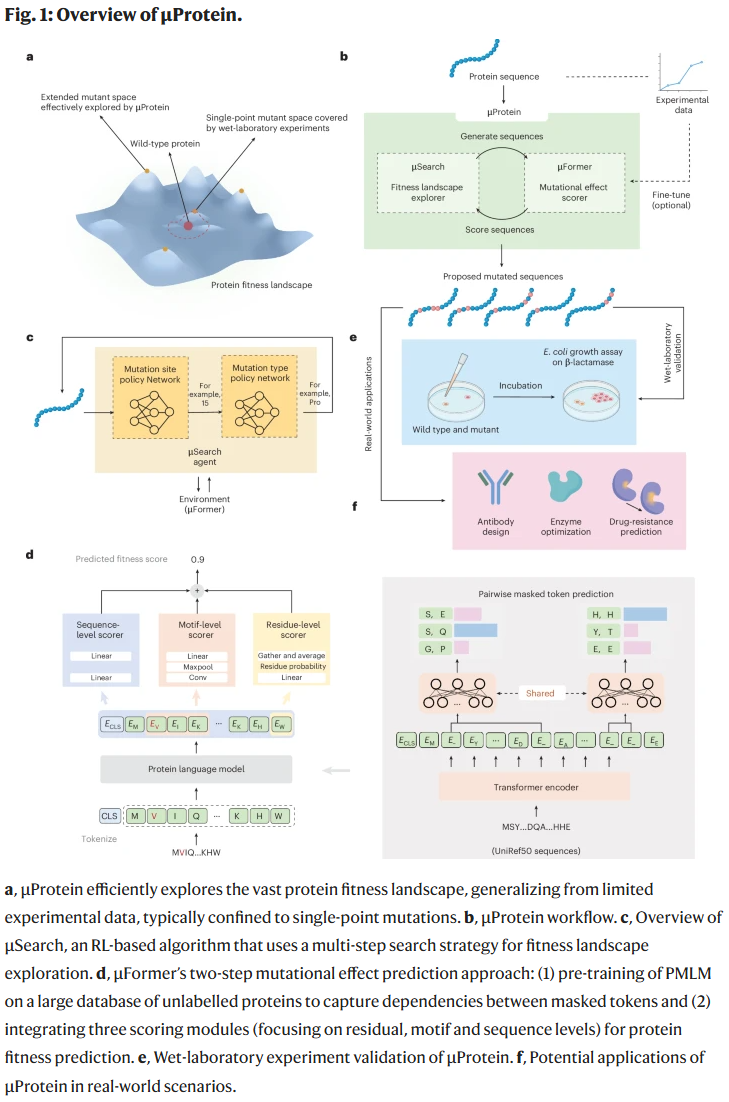
μFormer 在突变效应预测中的表现
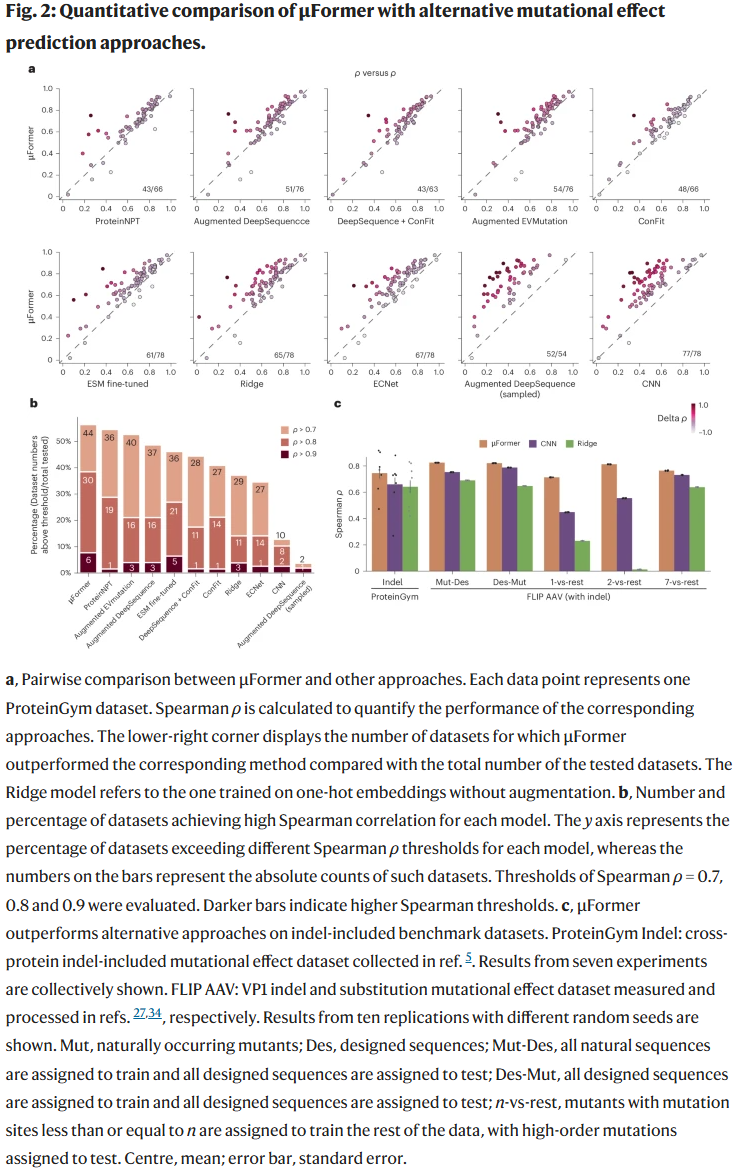
在 78 个 DMS 数据集上,μFormer 的表现优于 16 种对比方法。在超过一半的数据集中,其预测结果与真实适应度的相关性超过 0.7,并在部分数据集中超过 0.9,显示出其在不同蛋白质与功能类型中的稳健性。尤其在包含插入/缺失突变的任务中,μFormer 依然展现最佳性能,凸显其作为预测先知的广泛适用性。
表观遗传效应的捕捉
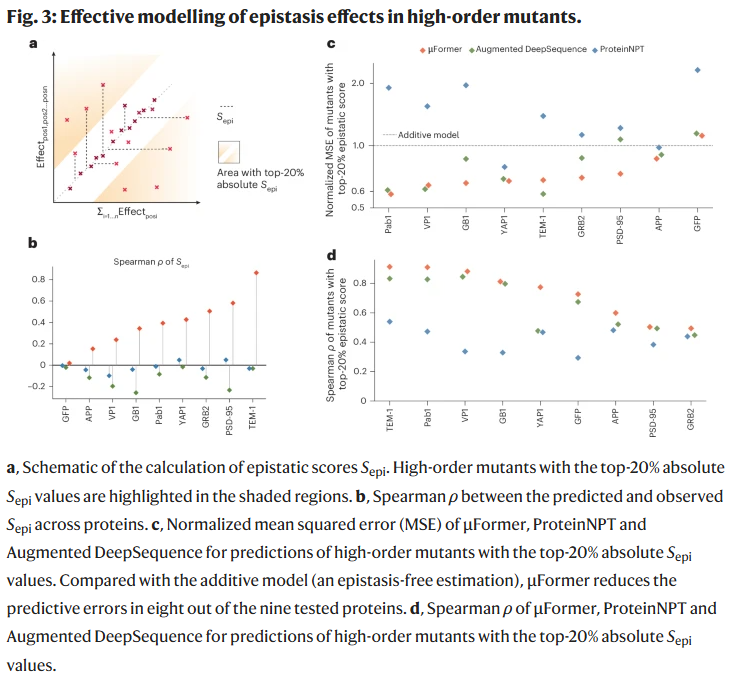
研究人员进一步验证了 μFormer 在预测多点突变时是否能够捕捉非加性效应。结果显示,其预测的表观遗传得分与真实值高度相关,并在大多数情况下显著降低了误差。这表明 μFormer 不仅能建模单点突变效应的累积,还能学习到复杂的残基交互关系。
高功能突变体的识别
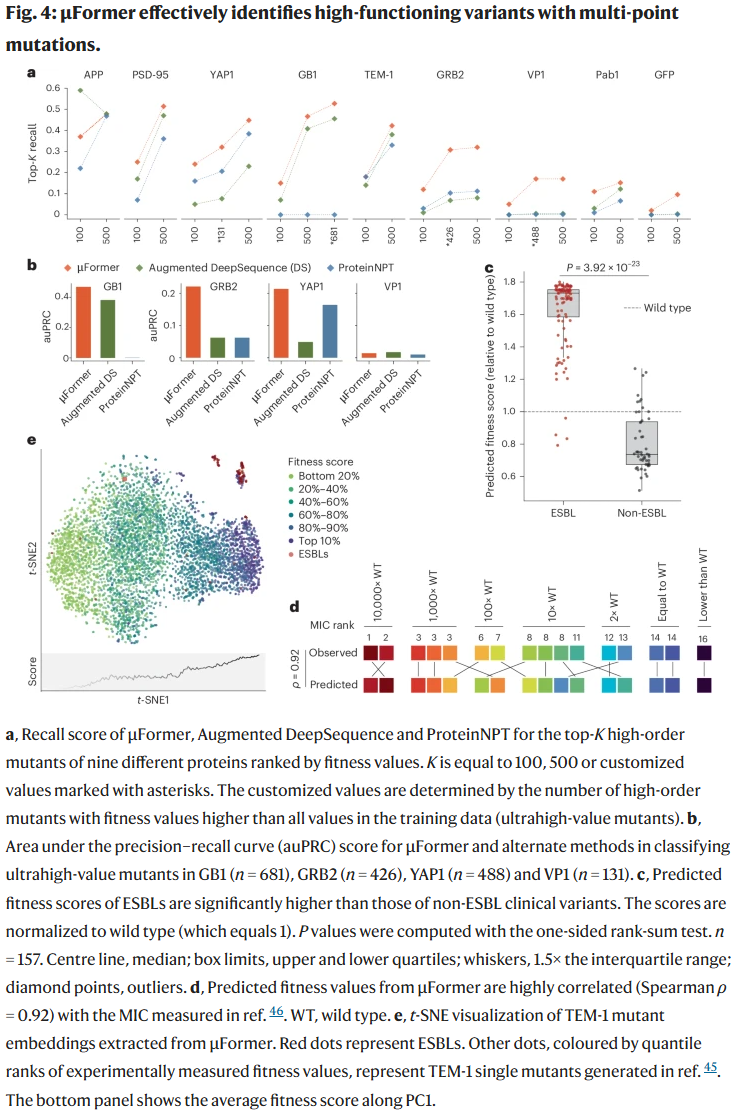
在多个高阶突变数据集中,μFormer 能有效预测表现最优的突变体。在 TEM-1 β-内酰胺酶的案例中,μFormer 成功优先排序了临床上已知的广谱耐药突变体(ESBLs),并与最小抑制浓度实验结果高度一致。这进一步验证了其实际应用价值。
μSearch 的探索能力
在多个适应度景观模拟任务中,μSearch 相比其他八种主流探索算法展现出更高的样本效率和搜索效果。在 TEM-1–头孢噻肟系统的实验中,μSearch 能够在较少的查询次数内识别到其他方法无法达到的高适应度序列,证明其在复杂景观中的强大导航能力。
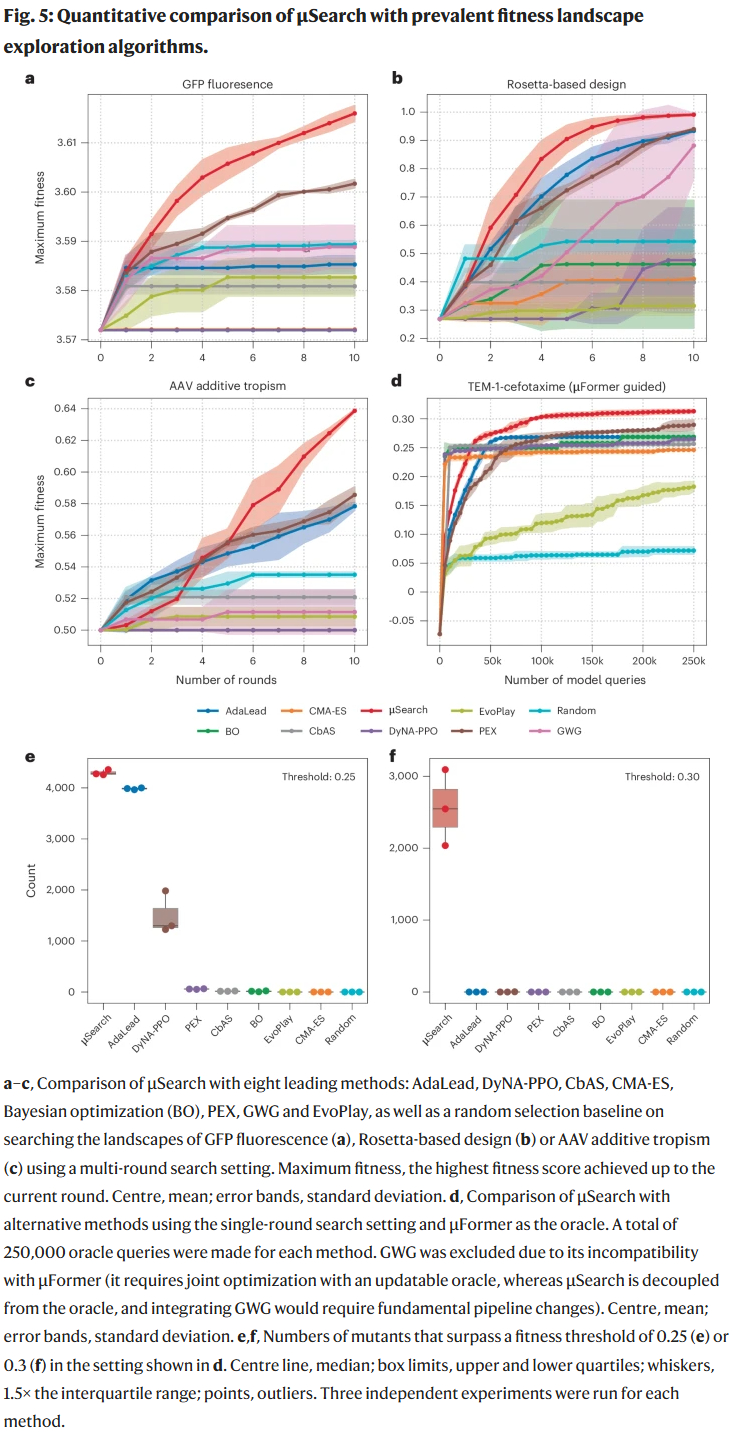
实验验证
研究人员在实验中设计并筛选了 200 个 RL(强化学习)生成的 TEM-1 突变体,结果有 47 个在头孢噻肟条件下的生长表现超过野生型,大幅优于随机生成的对照组。特别是双突变组合 G236S;T261V 的活性甚至超过了此前知名的四突变体,展示了 RL 设计突变体在突破自然进化局限上的潜力。
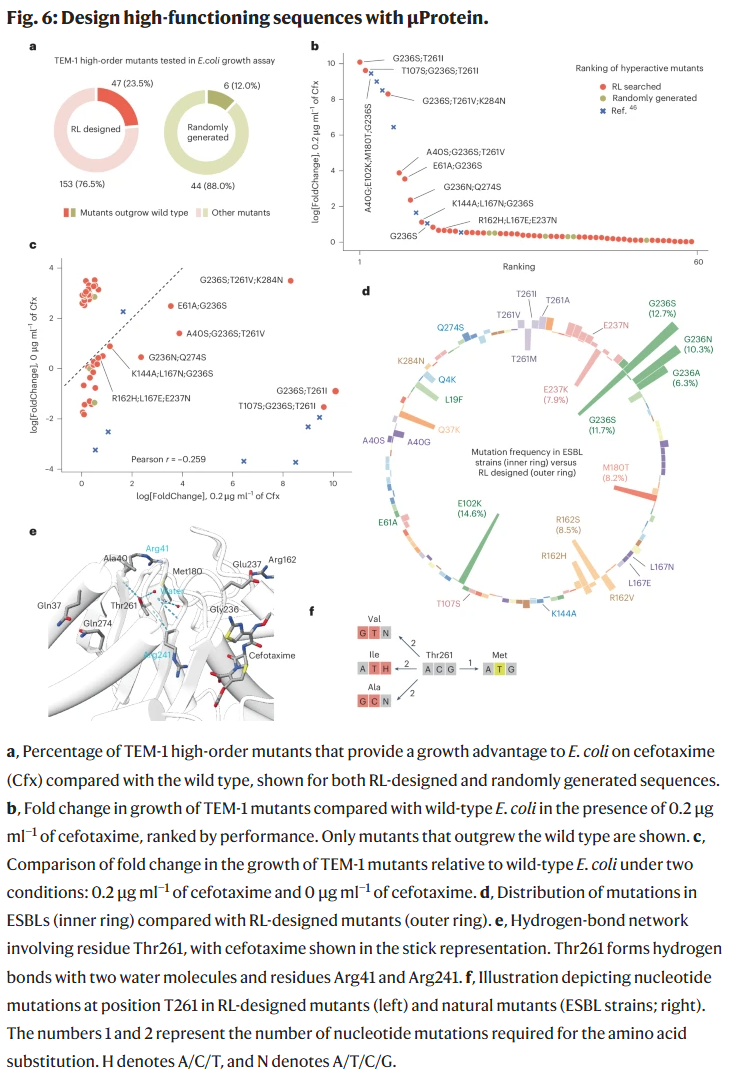
讨论
μProtein 框架通过结合深度学习与强化学习,实现了对蛋白质适应度景观的有效建模与探索。μFormer 提供高精度预测,μSearch 高效搜索突变空间,两者共同使得在有限实验数据条件下,依然能够发现具有高功能的蛋白质变体。
与传统方法相比,μProtein 在处理多点突变、插入/缺失突变等复杂场景中表现突出,并在实际实验中成功发现高功能变体,验证了其可行性。尽管如此,研究人员也指出,模型在不同任务中表现存在差异,未来可通过引入三维结构数据、丰富的环境变量以及迭代式实验反馈进一步提升性能。
总体而言,μProtein 为蛋白质工程提供了一种高效、稳健的新范式,为药物研发、抗体设计、耐药突变预测等应用开辟了新的可能性。
整理 | DrugOne团队
参考资料
Sun, H., He, L., Deng, P. et al. Accelerating protein engineering with fitness landscape modelling and reinforcement learning. Nat Mach Intell (2025).
https://doi.org/10.1038/s42256-025-01103-w

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢