通过这本实用、以项目为导向的指南,弥合现代机器学习与实际生物学之间的差距。无论你的背景是生物学、软件工程还是数据科学,Deep Learning for Biology 都能为你提供开发深度学习模型来处理各种生物学问题的工具。
作者 Charles Ravarani 和 Natasha Latysheva 将带你通过实际项目,将深度学习应用于 DNA、蛋白质、生物网络、医学图像和显微镜等领域。每一章都是一个独立的迷你项目,提供逐步解释,教你如何使用真实的生物学数据训练和解释深度学习模型。
为实际生物学问题(如基因调控、蛋白质功能预测、药物相互作用和癌症检测)构建模型。
应用卷积神经网络、Transformer、图神经网络和自动编码器等架构。
使用 Python 和交互式笔记本进行实践学习。
培养超越生物学范畴的问题解决直觉。
无论你是探索新方法、转向计算生物学领域,还是希望理解你所在领域的机器学习,这本书都为你提供了一条清晰且易于接近的路径。

年份:2025
出版社:O'Reilly Media, Inc.
下载地址:
链接: https://pan.baidu.com/s/1Xn9cZDVv7mn1jnhwTCKOsw?pwd=7hc4
书籍汇总:
链接: https://pan.baidu.com/s/1FFw_24YdJIUfLGunRGT_7g?pwd=9at9
链接: https://pan.baidu.com/s/1wp1sxh_p5Cv9dI5OpBaSCg?pwd=2arp
本书将带你领略生物学与深度学习之间引人入胜的交汇。本书的目标读者既包括渴望掌握计算技能的生物学家,也包括渴望将专业知识应用于生物学问题的计算实践者。这种学科融合正在改变生物技术和医学,并有望成为整个生命科学领域的基础。
本书的内容定位为入门级,引导您从基础知识到更中级的概念。我们力求在实用的代码示例和清晰的解释之间取得平衡,使新术语和新概念易于理解。本书会尽早并频繁地提供真实的 Python 代码,帮助您培养动手实践的直觉。虽然深度学习是一个强大的工具,但它并非万能的解决方案——我们强调在深入建模之前,理解数据并仔细构思问题的重要性。我们鼓励您从简单的开始,构建模块化且可调试的代码,并仅在有明确用途的情况下添加复杂性。
虽然本书面向初学者,但每一章都以上一章为基础,构建了一个实用的、端到端的工作流程,用于将机器学习应用于生物数据。我们的目标是为您提供足够强大的工具,以解决实际问题,并足够灵活地适应您自己的研究问题。例如,在最后一章中,我们重现了最近发表在《自然方法》杂志上的一篇论文的关键结果,该论文利用深度学习揭示了显微镜图像中的空间蛋白质模式。
这个领域发展速度惊人——数据库不断变化,数据集不断更新,模型架构也层出不穷。你将学习的核心思想——如何构建项目、准备数据、构建和评估模型,以及将预测与生物学问题联系起来——无论框架或趋势如何,都将始终有用。
我们始终使用 JAX 和 Flax 深度学习生态系统,帮助您使用机器学习研究中越来越流行的工具构建高效、灵活的模型。
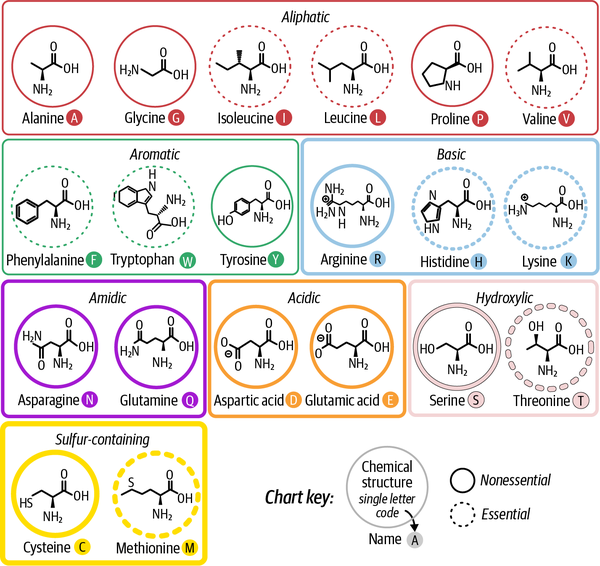
该图表显示了生物体中发现的 20 种标准氨基酸的化学结构,按生化相似性分组,按侧链特性(例如酸性、碱性、极性、非极性)进行颜色编码,并标注了它们的名称、单字母和三字母代码以及示例 DNA 密码子(编码该氨基酸的三联体 DNA 碱基)。
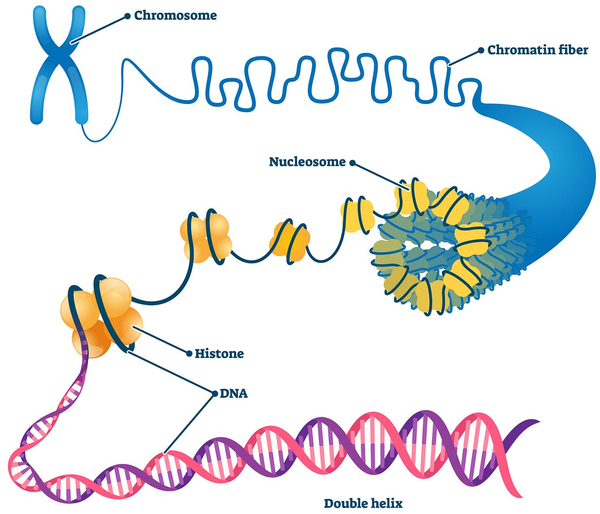
DNA以多层结构包裹在细胞核中。从双螺旋结构开始,它首先缠绕组蛋白形成核小体(串珠状结构),核小体折叠成染色质纤维,最终形成染色体。
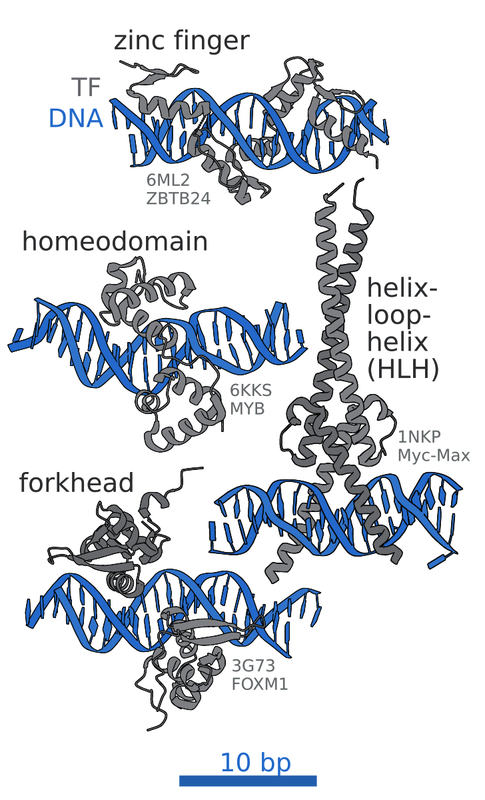
晶体学结构展示了不同类型的转录因子结合结构域如何与DNA相互作用。每个灰色结构代表一个不同的转录因子结合结构域——锌指结构域、同源结构域、螺旋-环-螺旋结构域和叉头结构域——与双链DNA分子结合。这些蛋白质片段通过这些序列形成的物理形状识别特定的DNA基序。就像钥匙插入锁一样,蛋白质的结构与其结合位点的DNA形状相辅相成。此处展示的是来自蛋白质数据库(PDB:6ML2、6KKS、1NKP、3G73)的实际解析结构。
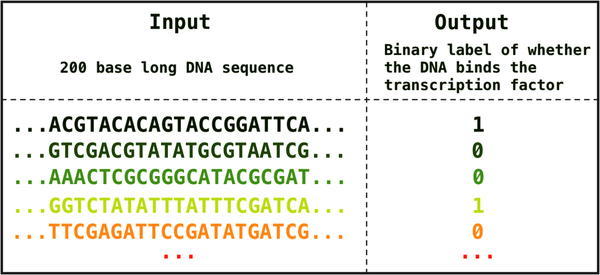
输入数据集由 DNA 序列组成,每个序列长 200 个碱基,并带有一个相关的二进制标签,指示蛋白质 CTCF 是否与其结合。


GraphSAGE 代表 Graph SAmple 和 AggreGatE,代表其两个主要步骤:(1) 对节点的邻居进行采样;(2) 聚合它们的特征以生成嵌入。这些嵌入可用于下游任务,例如 (3) 预测图中的节点属性或关系。
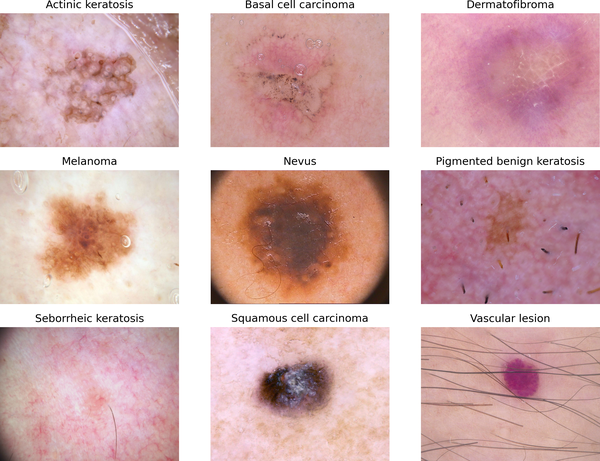
网格显示本章用于分类的数据集中各种类型的皮肤病变,包括良性和恶性病变。
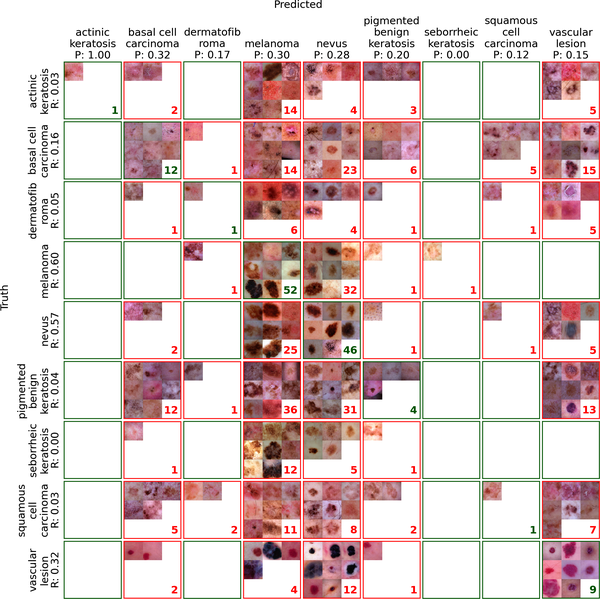
SimpleCnn 模型在九类皮肤病变分类任务中的混淆矩阵。行表示真实标签,列表示预测标签。对角线单元格表示正确预测;非对角线单元格表示错误分类。每个单元格还包含用于定性检验的示例验证图像,右下角显示预测数量。每个类别的准确率 (P:) 和召回率 (R:) 得分均按类别提供。
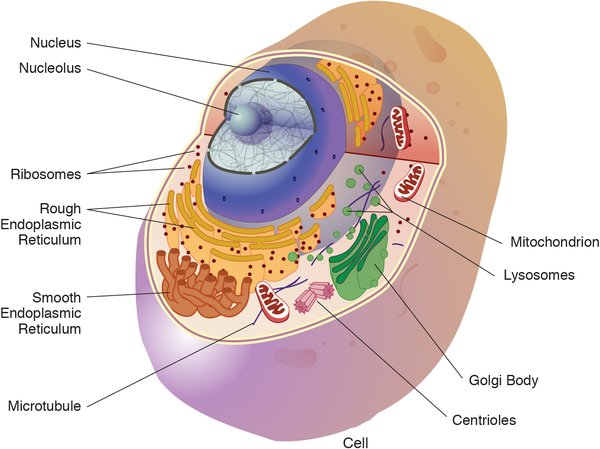
真核细胞内复杂组织的视觉呈现。不同的细胞器将细胞划分成不同的区域,这些区域分别执行不同的专门功能(插图来自美国国立卫生研究院)。
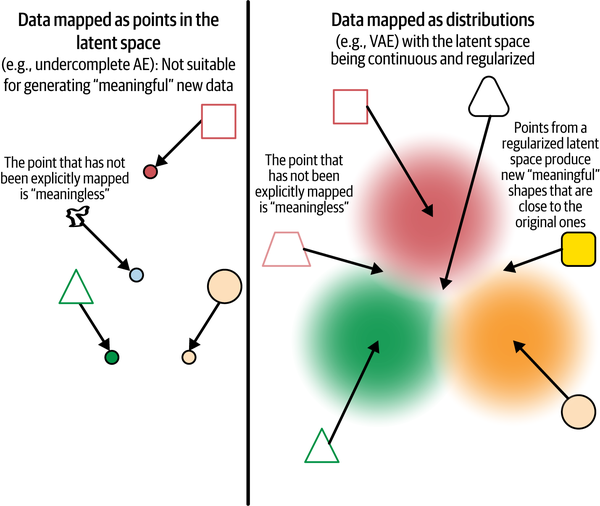
这是思考正则化连续潜在空间的一种直观方法。在标准自编码器中,点是离散映射的,可能无法进行有意义的泛化。在变分自编码器中,点是从平滑分布中采样的,从而实现有意义的插值和生成采样。
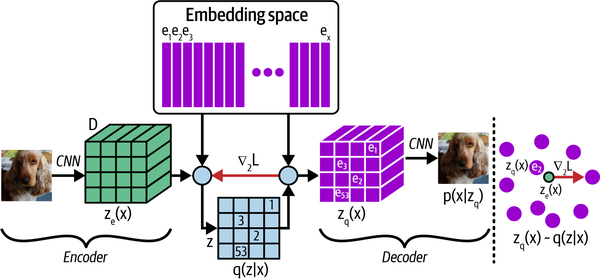
基于 van den Oord 等人 (2017) 的研究,VQ-VAE 中主要组件的说明。

OpenCell 数据库中 ACTB(β-肌动蛋白)蛋白质的示例条目。每个蛋白质都有一个详细的摘要页面,其中包含其标识符(例如 UniProt ID)、序列信息、表达水平(细胞中基因和蛋白质的拷贝数)以及亚细胞定位注释。右侧是荧光显微镜图像,显示 ACTB 定位(灰色)以及核染色(蓝色)。这些多通道图像与蛋白质特定的元数据一起构成了本章使用的数据集的基础。
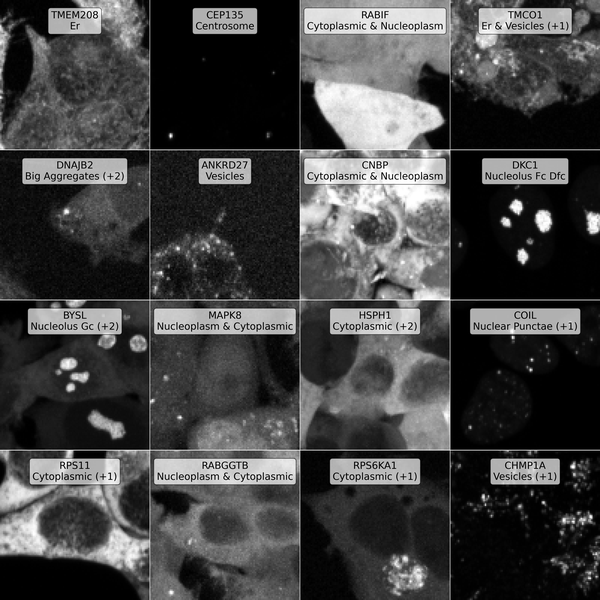
随机帧子集的图。每帧均给出了蛋白质符号及其主要定位(数字表示测量到的额外次要定位)。



微信群


内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢