DRUGONE
蛋白质复合物结构预测对于理解生物活动与推动药物研发至关重要。尽管已有多种实验方法能提供结构信息,但通常零散或不完整,因此亟需一种通用工具来高效整合有限实验数据。研究人员提出了 GRASP,能够灵活地融合不同类型的实验约束,并在模拟与真实数据中均表现优于现有方法。尤其在抗原–抗体复合物预测中,GRASP 在结合深度突变扫描或共价标记约束时甚至超越了 AlphaFold3。更重要的是,GRASP 支持多种约束的联合建模,并展示了在近细胞条件下解析线粒体蛋白互作网络的潜力。

蛋白质–蛋白质相互作用在多种生物过程中至关重要,但已知氨基酸序列与解析结构之间仍存在巨大差距。现有预测方法如 AlphaFold-Multimer、AlphaFold3、ESMFold 和 OmegaFold 在复合物预测中精度有限。
实验方法可提供有价值但稀疏的信息:
残基对约束(RPRs):来源于交联质谱或 NMR 实验,提供残基间的距离信息。
界面约束(IRs):如差异共价标记、化学位移扰动、深度突变扫描等方法识别界面残基。
此外,冷冻电镜断层成像、溶液 NMR、细胞内交联等方法能提供更贴近生理状态的互作数据。如何有效整合这些不同来源的信息,是当前建模的重要挑战。
结果
GRASP 的建模框架
GRASP 将 RPR 和 IR 同时整合进 AlphaFold-Multimer 框架:
RPR 作为图边特征引入,用于约束残基对距离。
IR 作为节点特征,标记潜在界面残基。
研究人员在模型中加入四个与约束相关的损失函数,并采用迭代噪声过滤策略以降低错误约束的影响。
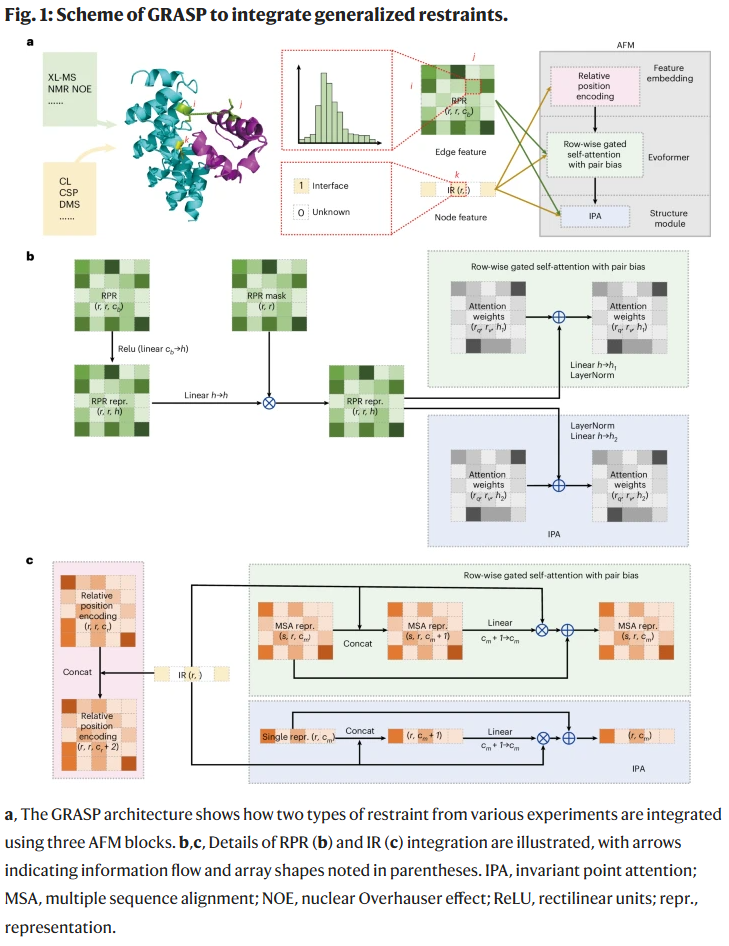
模拟数据上的验证
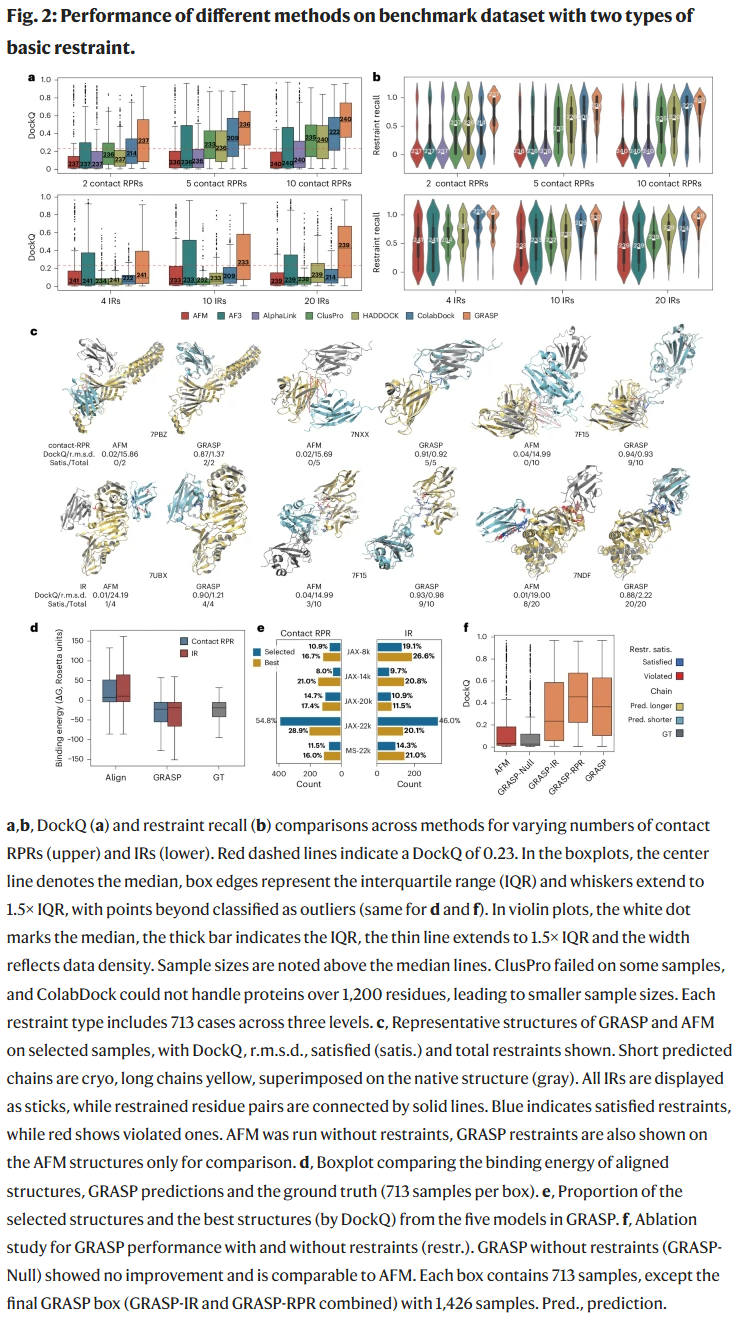
在包含 713 个界面的基准数据集上,GRASP 在不同数量的 RPR 与 IR 条件下均表现最佳。例如,仅需两个跨链接触约束,GRASP 就能将 DockQ 提升至 0.35,并使一半以上样本达到可接受水平。相比之下,AlphaLink、ClusPro、HADDOCK 和 ColabDock 整体表现不及 GRASP。
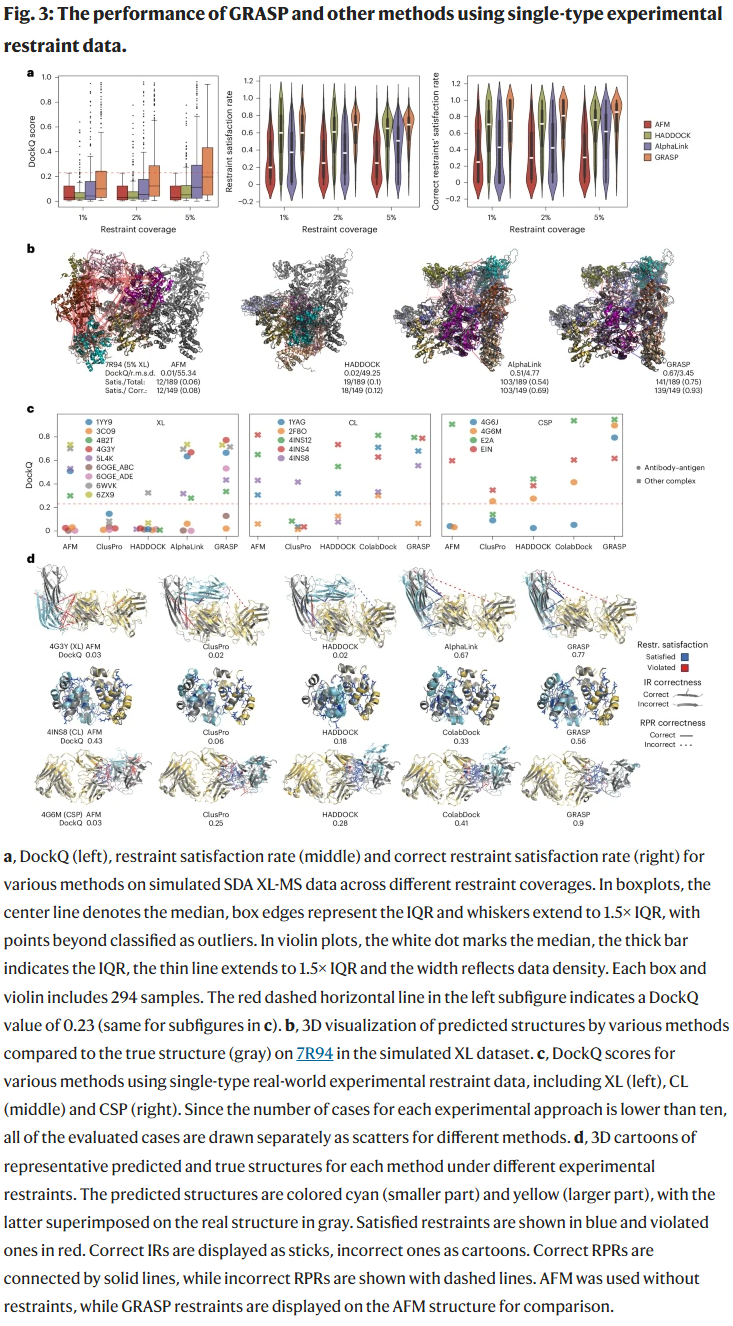
基于 XL、CL 与 CSP 约束的实验数据
研究人员进一步在交联质谱(XL)、共价标记(CL)和化学位移扰动(CSP)数据上评估 GRASP:
XL 数据:在覆盖率仅 1% 的情况下,GRASP 依然显著提升 DockQ,并在存在 20% 错误约束时保持稳健。
CL 数据:GRASP 平均 DockQ 高于其他方法,个别案例中仅 GRASP 超越了 AlphaFold-Multimer。
CSP 数据:在抗原–抗体复合物中,GRASP 表现远优于其他方法。
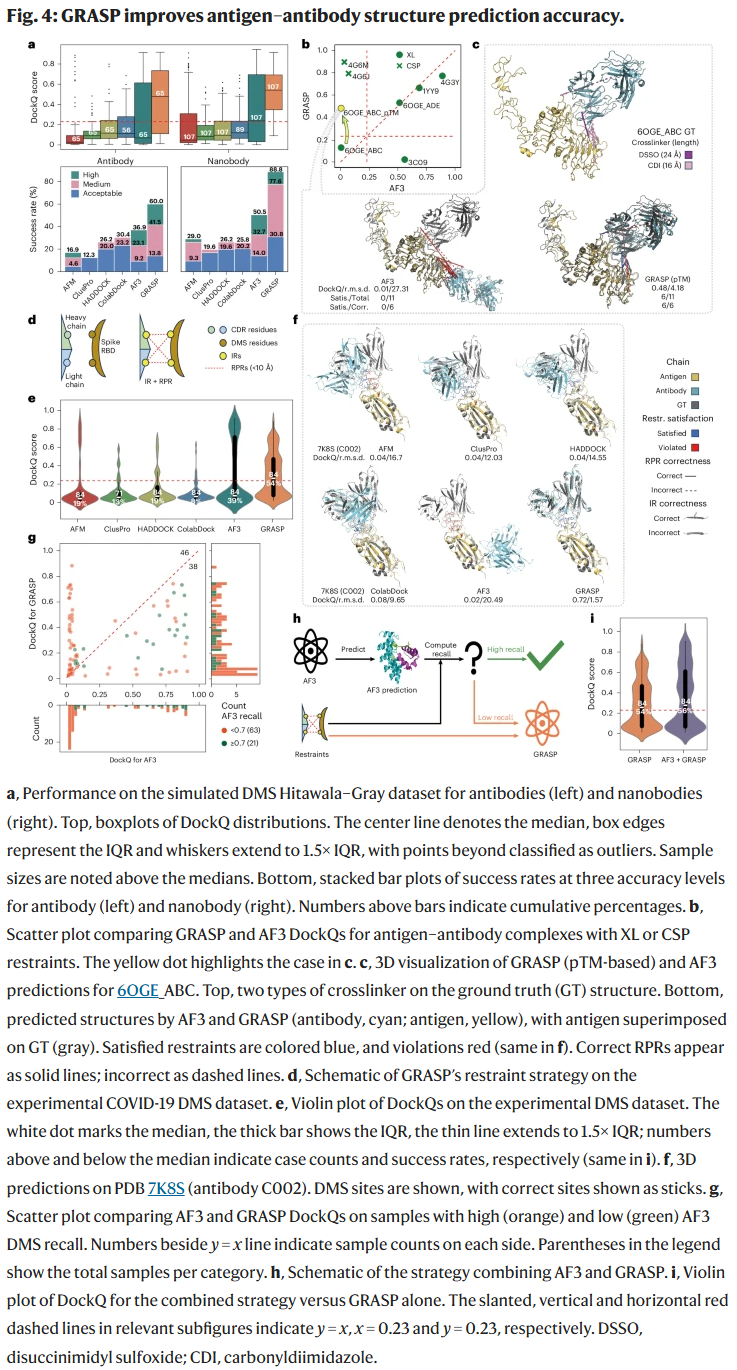
抗原–抗体复合物预测
抗原–抗体预测是最具挑战性的任务之一。GRASP 在模拟与真实的深度突变扫描数据集上均取得领先结果,成功率大幅超越 AlphaFold3。即使在部分约束不准确的情况下,GRASP 仍能保持较高的预测精度,并且对噪声具有较强的抵抗力。
多源约束的整合建模
研究人员展示了 GRASP 在多源实验数据下的应用:
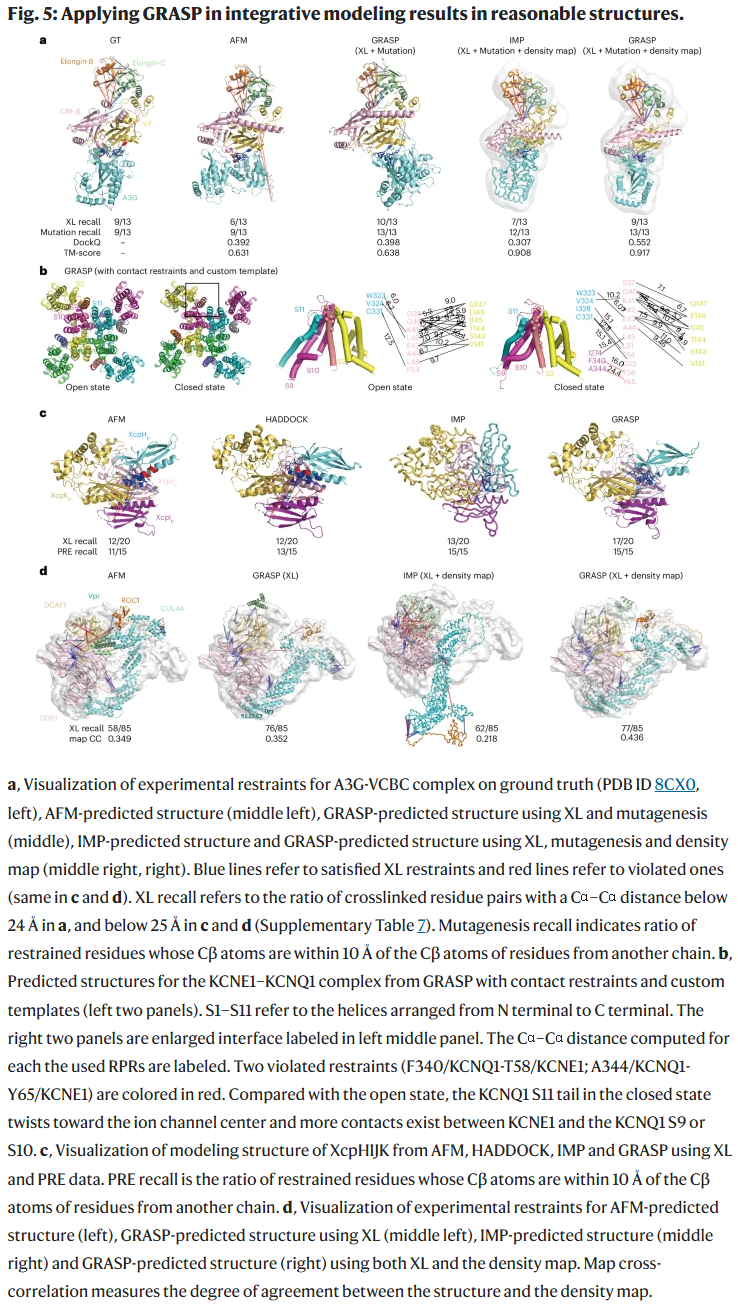
A3G–Vif–VCBC 复合物:整合 XL、突变数据与中等分辨率冷冻电镜图谱,GRASP 提供了比单一数据更合理的结合构象。
KCNQ1–KCNE1 复合物:通过二硫键交联和双突变分析,GRASP 成功区分了开放与关闭两种状态。
XcpHIJK 复合物:结合 XL 与 NMR 数据,GRASP 的预测同时满足大部分实验约束。
CRL4DCAF1–Vprmus 装配体:GRASP 融合 XL 与冷冻电镜密度图,得到的结果优于 AFM 与 IMP。
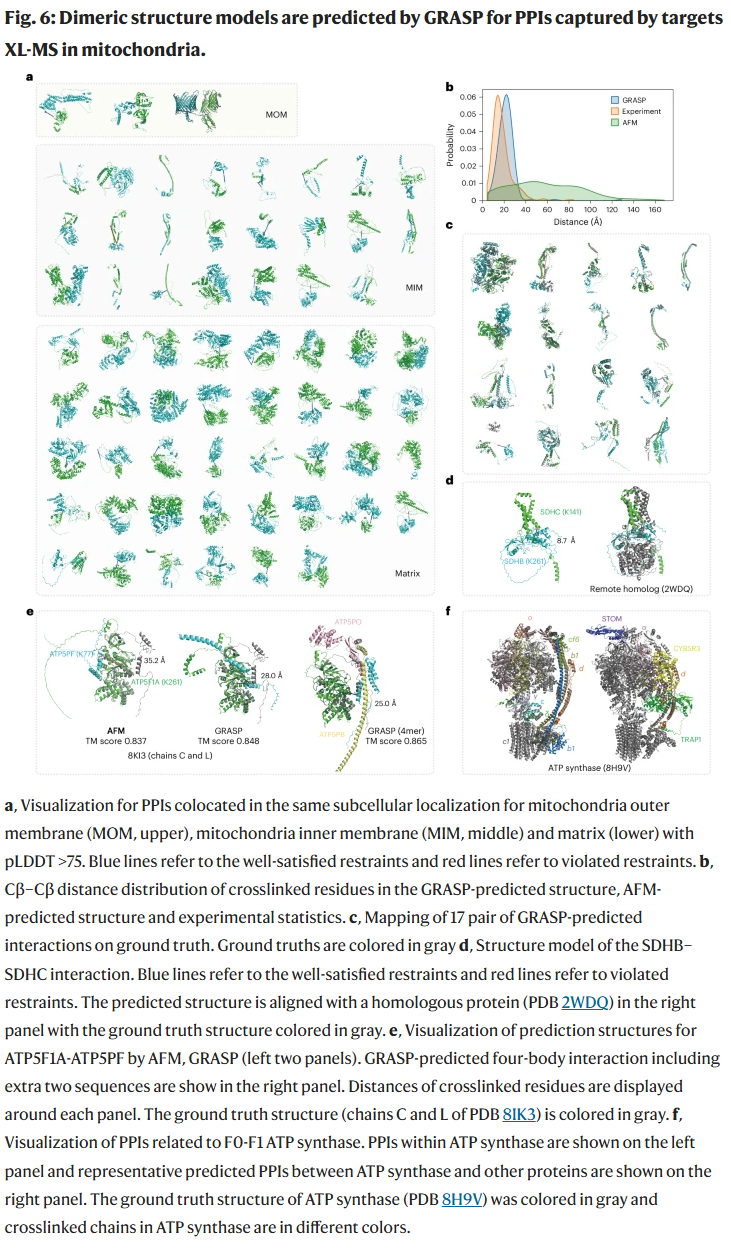
线粒体原位互作建模
研究人员利用大规模线粒体交联质谱数据,将其转化为约束输入 GRASP,预测了 144 对蛋白质互作。结果显示,GRASP 的预测与实验数据分布高度一致,并能在多个已知真实结构中得到验证。部分互作如 ATP 合酶的亚基互作,预测结果与同源结构高度吻合。
讨论
研究人员提出的 GRASP 是一种通用框架,能够在蛋白质复合物结构预测中高效整合多种实验约束。与现有方法相比,GRASP 的优势在于:
同时处理残基对约束与界面约束;
支持多类型实验数据的联合建模;
引入额外损失函数与迭代噪声过滤以增强稳健性;
在抗原–抗体预测和复杂系统的整合建模中表现优异。
当前 GRASP 的约束形式主要局限于 RPR 与 IR,但已有初步尝试将冷冻电镜密度图转化为约束。未来,结合小角 X 射线散射、冷冻电镜等更多实验数据,将进一步拓展其应用范围。
在 AlphaFold 之后的时代,研究人员愈发关注复杂的蛋白质互作建模。GRASP 的提出不仅为解析大规模互作网络提供了工具,也为跨模态、跨层次的整合建模开辟了新途径。
整理 | DrugOne团队
参考资料
Xie, Y., Zhang, C., Li, S. et al. Integrating diverse experimental information to assist protein complex structure prediction by GRASP. Nat Methods (2025).
https://doi.org/10.1038/s41592-025-02820-1

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢