DRUGONE
医疗决策依赖于对患者既往和当前健康状态的理解,以预测并改变其未来病程。研究人员开发了 Delphi-2M,一种基于生成式预训练 Transformer(GPT)改造的模型,能够在个体既往病史条件下预测 1000 多种疾病的发生率,并生成个体未来健康轨迹的合成样本。该模型在英国生物样本库(40 万人)上训练,在丹麦国家队列(190 万人)上无需参数调整即可外部验证,预测准确度可与单病种模型媲美。Delphi-2M 还能揭示共病聚类和时间依赖性,对个体化健康风险评估和精准医疗具有潜在应用价值。

人类疾病进程伴随急性发作、慢性损伤和多病共存,受生活方式、遗传特质及社会经济因素影响。传统风险预测模型多针对单一疾病,无法涵盖 ICD-10 一级编码下的 1000 多种疾病。随着全球人口老龄化和慢性病负担增加,急需能同时建模多病进程的工具。
Transformer 架构在自然语言处理中展现了强大的序列建模能力,与疾病演进过程类似:均需利用既往事件预测未来。虽然已有基于 BERT 或 Transformer 的电子健康记录预测尝试,但全面的多病种生成式建模尚未得到系统评估。Delphi-2M 填补了这一空白。
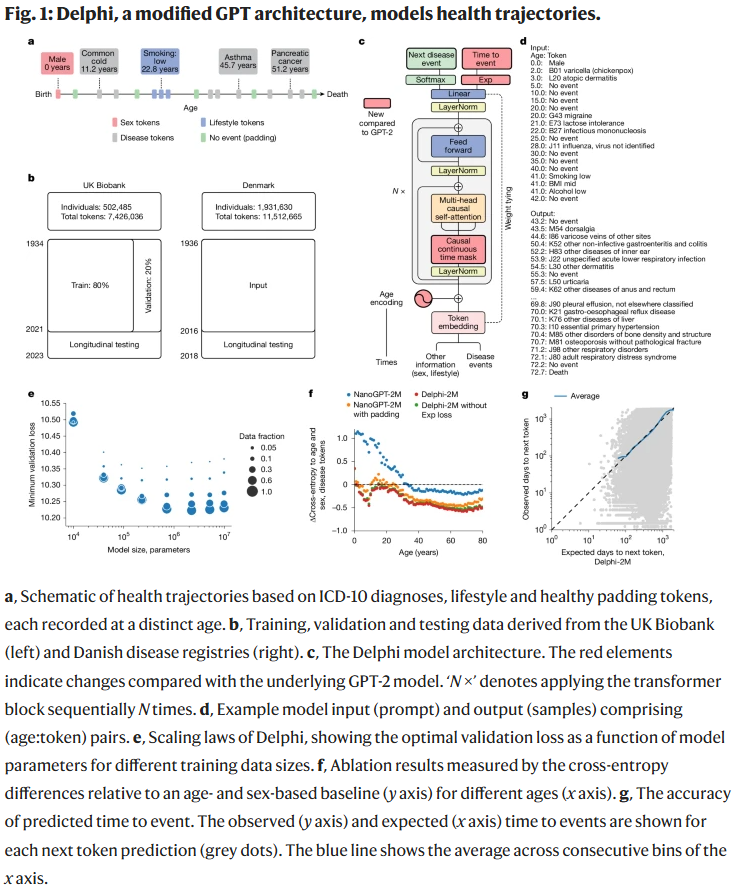
结果
多病种预测性能
Delphi-2M 在内部验证中,对 97% 的诊断预测 AUC > 0.5,平均 AUC 约 0.76。对于死亡,预测最为准确(AUC=0.97)。模型性能在长期预测(10 年)中仍保持稳定(AUC≈0.70)。在部分疾病上,其预测性能与临床常用风险评分相当或更优。
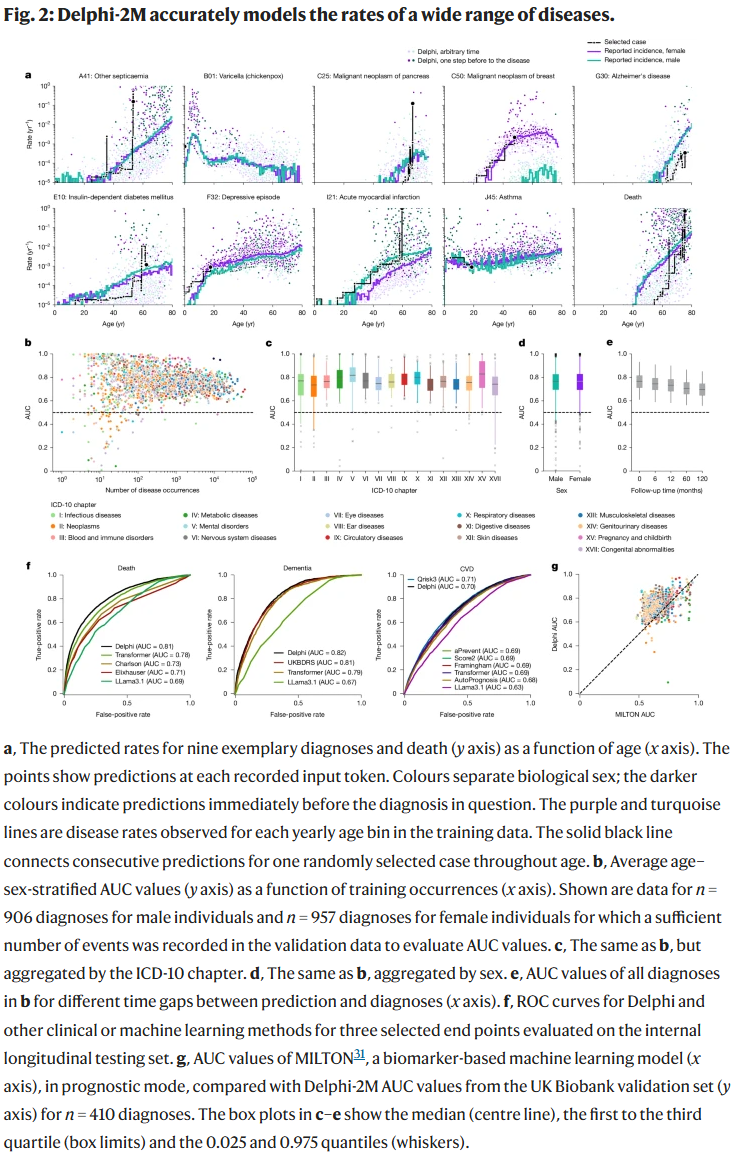
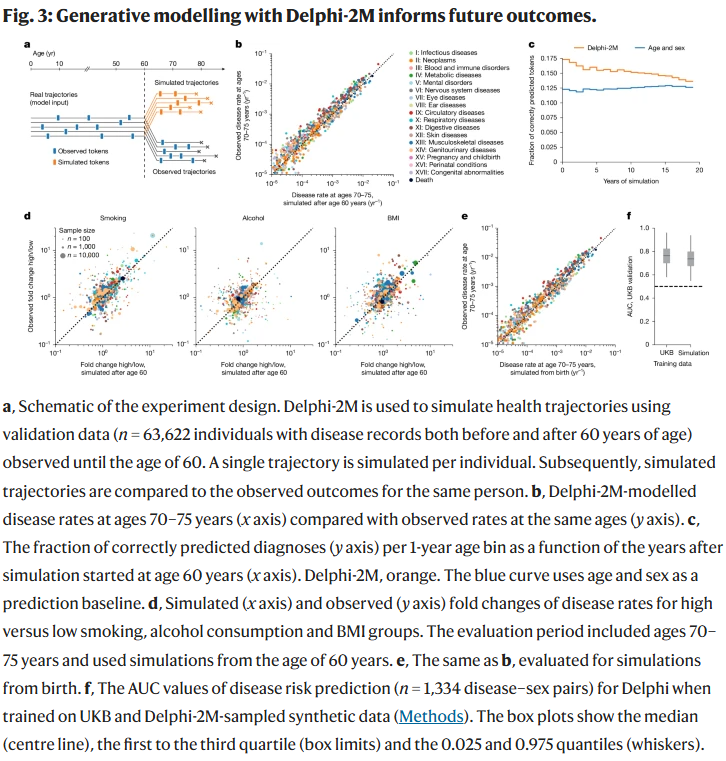
未来健康轨迹生成
Delphi-2M 能够基于个体既往病史生成未来疾病轨迹,20 年尺度上仍能保持合理的疾病分布趋势。模拟结果在吸烟、饮酒、BMI 等人群亚组中均能重现疾病风险差异。
此外,研究人员用完全合成数据训练的模型在真实验证集上的表现仅比原始模型低 3 个百分点,显示合成数据可用于保护隐私的同时保留统计特征。
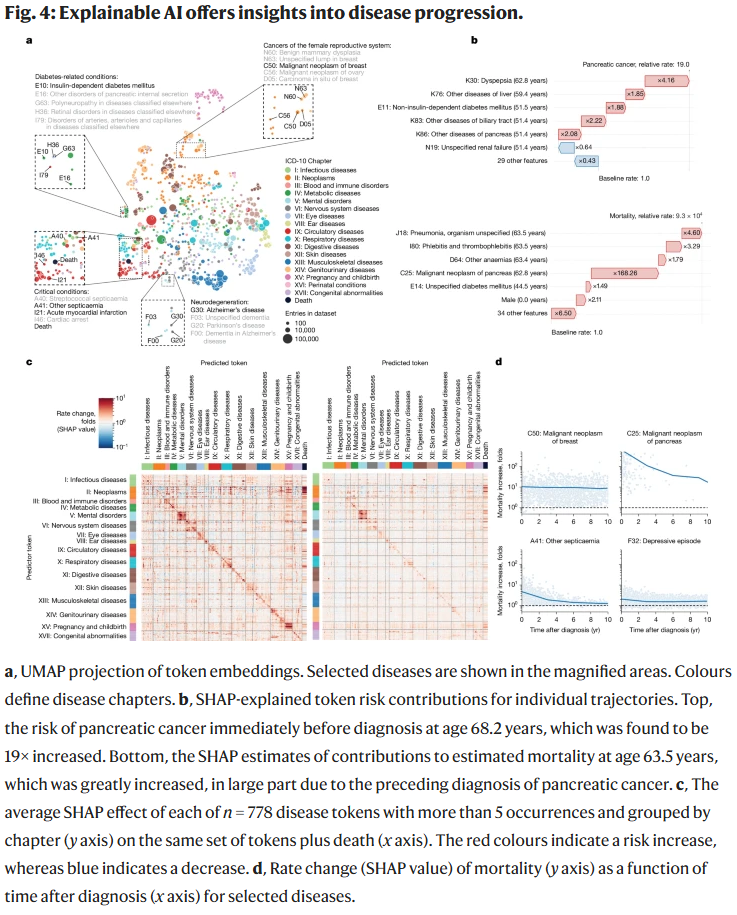
可解释性分析
利用 SHAP 方法,研究人员量化了各个既往疾病对未来风险的影响。例如消化道疾病显著增加胰腺癌风险,而胰腺癌进一步极大提高死亡风险。UMAP 降维结果显示,疾病编码在嵌入空间中按 ICD-10 章节聚类,但也揭示了跨章节的共病模式,如糖尿病与视网膜病变和神经病变的关联。
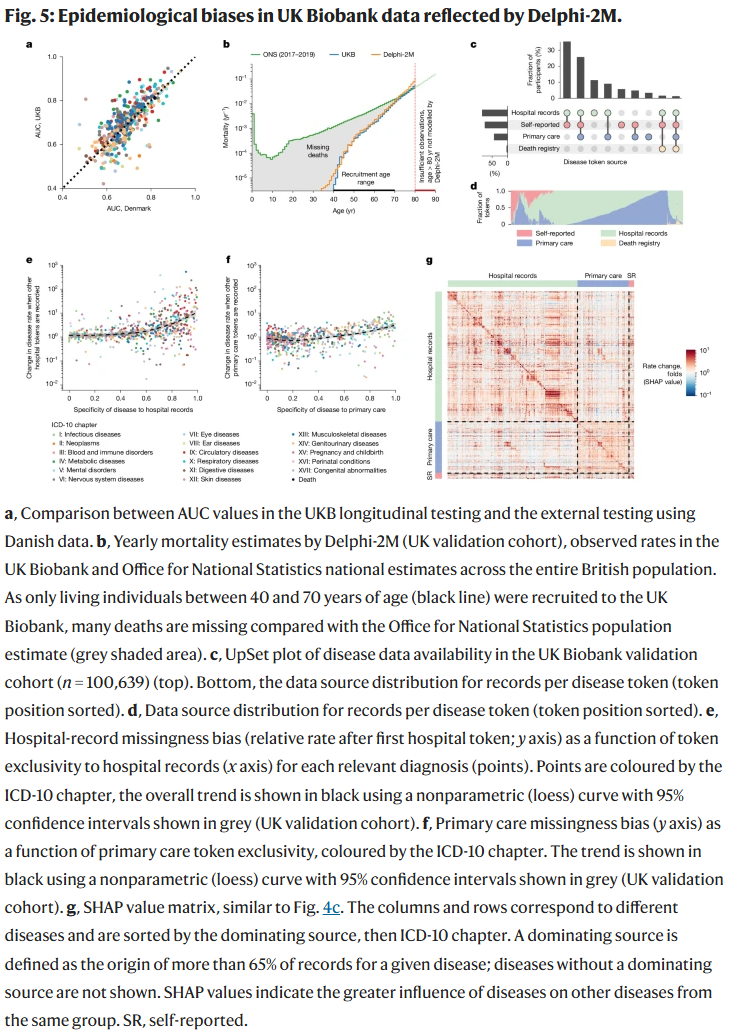
外部验证与偏倚
在丹麦队列上,Delphi-2M 平均 AUC 为 0.67,仅略低于英国数据,显示其跨国适用性。然而,模型也学习到英国生物样本库特有的偏倚:
招募偏倚:受试者以 40–70 岁健康志愿者为主,导致死亡率低估。
数据源偏倚:医院数据主要包含严重疾病,缺失导致预测偏差。
这些结果提示在异质人群中使用模型时需谨慎解读。
讨论
Delphi-2M 展示了 Transformer 在疾病自然史建模中的潜力:
优势:多病种联合预测、长时间尺度的疾病轨迹生成、隐私友好的合成数据生成。
启示:揭示共病模式和时间依赖性,为多病种关联研究和健康风险分层提供新工具。
局限:存在健康志愿者偏倚、数据缺失和族群代表性不足;预测结果应谨慎解读为因果关系。
未来,研究人员建议扩展至多模态输入(基因组、代谢组、影像学、可穿戴设备数据等),并探索 Delphi 与通用 LLM 的结合,用于临床决策支持和公共卫生规划。
整理 | DrugOne团队
参考资料
Shmatko, A., Jung, A.W., Gaurav, K. et al. Learning the natural history of human disease with generative transformers. Nature (2025).
https://doi.org/10.1038/s41586-025-09529-3

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢