DRUGONE
随着蛋白质数据库规模的快速增长,对更快、更敏感的同源性搜索工具的需求愈发迫切。研究人员开发了 GPU 加速版 MMseqs2 (MMseqs2-GPU),在单一蛋白搜索中比基于 128 核 CPU 的方法快 6 倍,在大规模批量任务中,使用 8 张 GPU 时可实现 2.4 倍的成本效率提升。MMseqs2-GPU 能显著加速结构预测与比对任务,例如在 ColabFold 中的多序列比对生成较 AlphaFold2 标准流程快 31.8 倍,在 Foldseek 中的结构搜索快 4–27 倍。该工具已开源,面向全球科研人员。
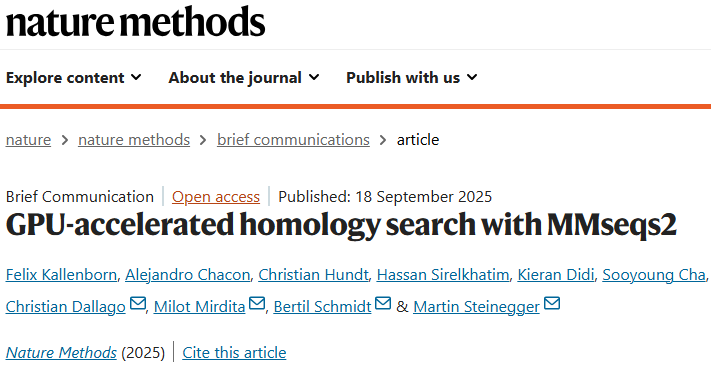
蛋白质同源性搜索是理解功能与结构的核心环节。传统的 Smith–Waterman–Gotoh 动态规划算法能保证最优比对,但计算代价极高。为提高效率,BLAST、PSI-BLAST、MMseqs2 与 DIAMOND 等工具采用启发式方法;HMMER 与 HHblits 则使用更敏感但计算代价高的基于动态规划的筛选。然而,即便有 CPU 并行化优化和 FPGA 加速器,面对超大规模数据库时仍然受限。GPU 的并行优势为加速同源性搜索提供了新的机会。研究人员将两类 GPU 加速算法集成到 MMseqs2 中:无缺口筛选与 基于 PSSM 的有缺口比对,在保持灵敏度的同时显著提高了速度和效率。
方法
算法设计
无缺口筛选 (Gapless filter):利用 GPU 将查询 PSSM 映射到矩阵列,参考序列映射到行,每一行并行处理,结合共享内存和 16 位数据表示,实现高吞吐率。
Smith–Waterman–Gotoh GPU 实现:在 CUDASW++4.0 的基础上修改,使其适应 PSSM 输入,采用波前模式处理动态规划依赖。
并行与优化
多线程分组与瓦片优化,用于处理不同长度的序列。
使用共享内存副本和 warp-shuffle 指令,减少访存冲突并加快线程间通信。
支持数据库分片与多 GPU 分布式计算。
通过数据库流式传输,实现对超大数据库的处理,避免 GPU 内存瓶颈。
结果
性能提升
在随机序列上,单张 L40S GPU 可达 13.5 TCUPS,速度提升 2.8 倍;8 张 GPU 可达 102 TCUPS,提升 21.4 倍。
在真实蛋白质序列上,速度进一步提升至单卡 18.4 倍,多卡 110 倍,超过以往 FPGA 和 GPU 加速方法两个数量级。
搜索敏感性
在保持 ROC1 ~0.40 的条件下,MMseqs2-GPU 与 CPU 版本具有相同敏感性。
迭代搜索可进一步提高灵敏度,三次迭代时 ROC1 接近 JackHMMER。
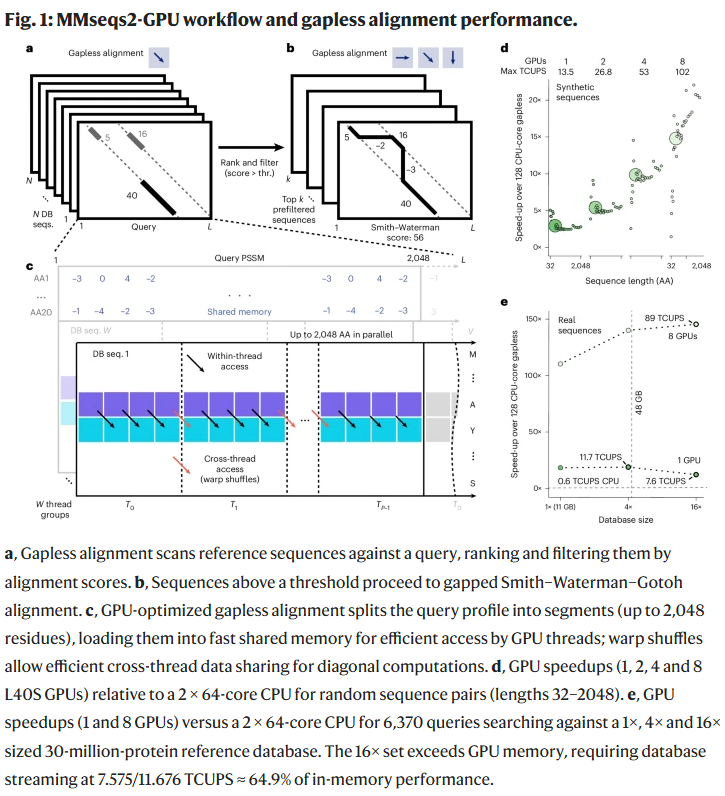
单查询与大批量性能
在单查询下,MMseqs2-GPU 比 JackHMMER 快 177 倍,比 BLAST 快 6.4 倍。
在 6370 个查询的批量任务中,8 张 GPU 下比 CPU 快 2.4 倍。
云计算成本比较显示,MMseqs2-GPU 是所有方法中最具性价比的方案。
结构预测与 Foldseek 搜索
在 ColabFold 流程中,MMseqs2-GPU 使多序列比对生成加速 176 倍,整体结构预测加速 31.8 倍,同时保持 TM-score (0.70 ± 0.05) 不变。
在 Foldseek 中,单卡 GPU 加速比 CPU 快 4 倍,8 卡提升至 27 倍,且灵敏度略有提升。
能效与内存优化
在单批次场景中,4 张 L40S GPU 的能效比 JackHMMER 高 80 倍,比 CPU MMseqs2 高 2.1 倍。
内存需求从每个残基 ~7 字节降低到 ~1 字节,支持数据库分布式存储与流式加载,进一步减少硬件限制。
讨论
MMseqs2-GPU 展示了 GPU 加速在蛋白质同源性搜索中的巨大潜力:
高速度与高效率:显著超越传统 CPU、GPU 及 FPGA 方法。
低成本与低门槛:云计算环境中具备最佳性价比,支持 ColabFold 等轻量化环境使用。
广泛适用性:可直接应用于结构预测、同源性检索、蛋白质语言模型、直系同源推断等多个场景。
局限性包括:
高灵敏度搜索仍需多次迭代,存在计算负担;
超长序列(>2000 aa)的比对仍需进一步优化;
部分 GPU 型号性能差异较大(如 A100 较 L40S 慢约 1.6 倍)。
未来方向:
融合时间动态数据与蛋白质语言模型,加速多序列比对与相似性学习;
扩展至大规模宏基因组数据库,加快新功能蛋白发现;
优化跨 GPU 集群的分布式架构,提升大规模任务的并行效率。
总结
MMseqs2-GPU 在保持高灵敏度的同时,大幅加速蛋白质同源性搜索、结构预测与比对任务,并显著降低能耗与成本。这一框架不仅为基础生物学研究提供了高效工具,也为药物研发、蛋白质工程和人工智能驱动的生物学探索提供了技术支撑。
整理 | DrugOne团队
参考资料
Kallenborn, F., Chacon, A., Hundt, C. et al. GPU-accelerated homology search with MMseqs2. Nat Methods (2025).
https://doi.org/10.1038/s41592-025-02819-8

内容为【DrugOne】公众号原创|转载请注明来源
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢