
我们来自TAILab(Trust & Application AI Lab),隶属于香港科技大学(广州)AI Thrust,由戴恩炎(EnyanDai)教授带领。团队聚焦于可解释性、可靠性以及AI在实际应用场景中的落地,涵盖图神经网络攻击与防御、可信图学习、多模态AI等方向。TAILab近年来积极探索AI驱动的分子建模、蛋白质结构表征和功能预测等前沿方向。
研究动机与问题
蛋白酶切是蛋白质加工、信号转导、免疫反应等中枢环节。实验测定切割位点(质谱、肽段实验)成本高、覆盖低;而数据库(如MEROPS)对酶的覆盖有限。目前已经有了很多对于酶切位点预测的工具,但这些工具往往局限于对单一酶类进行切割预测,并且难以泛化。本文核心问题:如何在多酶统一建模下,利用活性位点知识提升切割位点预测的准确性并泛化到未见酶?
模型架构
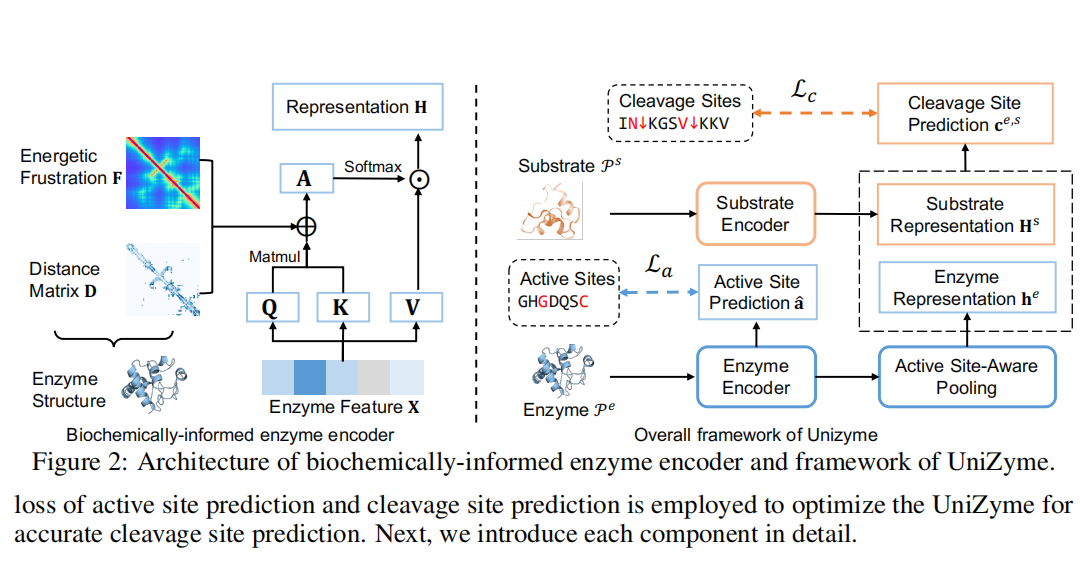
核心设计
生化先验酶编码器:将局部能量挫折(Frustration,基于AWSEM势能)与三维距离编码为注意力偏置,让注意力更关注“功能相关”的局部区域。
活性位点辅助任务:以活性位点预测作为辅助任务进行大规模预训练,并在主任务联合优化,提高酶的功能表征。
活性位点感知池化(ActiveSite-AwarePooling):用预测到的活性位点概率,学习池化权重,让酶全局表征更聚焦催化相关残基。
子序列局部判别:模拟真实切割窗口(~31aa),将局部底物表示与酶表示拼接判别是否发生切割。
模型首先通过生化先验酶编码器,把酶的结构输入结合局部能量挫折与三维距离矩阵,作为注意力偏置来突出功能相关区域。接着,模型在酶侧引入活性位点预测辅助任务,并利用活性位点感知池化聚焦关键残基,从而得到更精准的酶全局表征。底物则通过独立的编码器建模,最后与酶表征拼接,在联合优化的监督下完成切割位点预测。这种设计将活性位点知识显式融入表示学习,使得模型在跨酶统一建模与零样本泛化上具备优势。
实验结果:Supervised&Zero-Shot
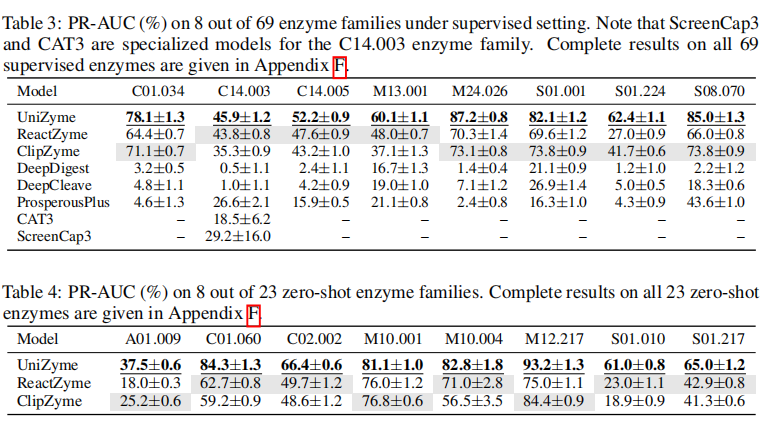
在监督设定下,UniZyme在大部分家族的PR-AUC提升10–30个百分点。
在零样本设定下,UniZyme的优势更明显,例如在C01.060家族中,UniZyme达到84.3%,而ReactZyme仅有62.7%,ClipZyme更低只有59.2%。
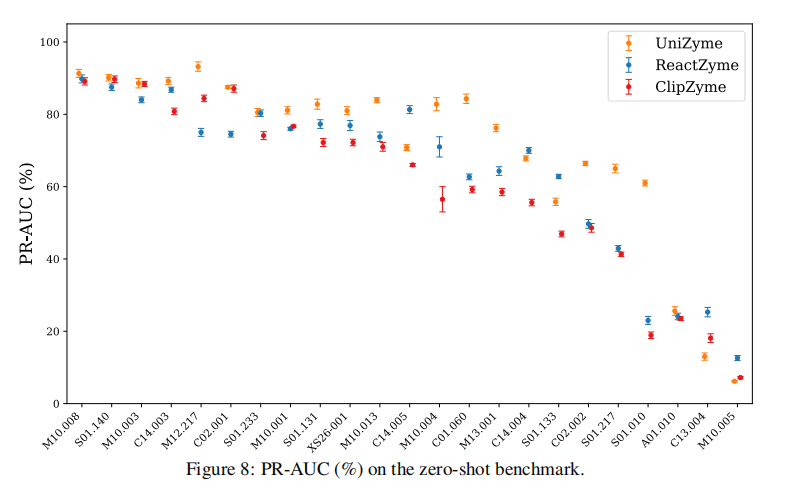
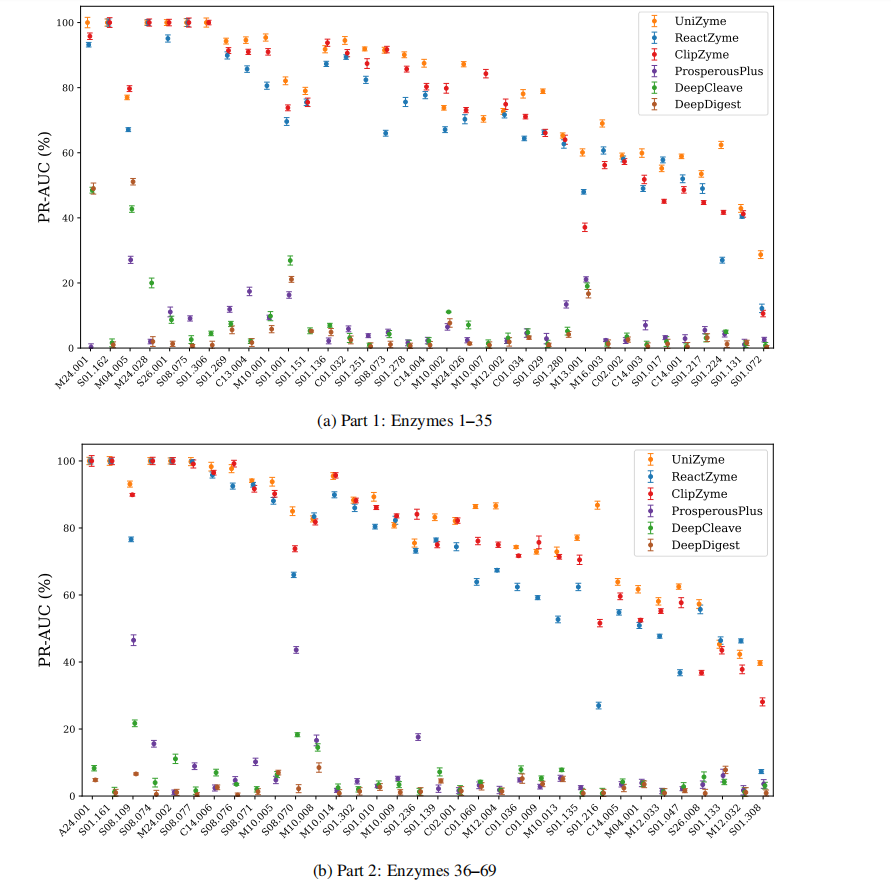
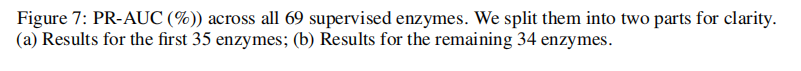
展示了所有69个Supervised和23个Zero-shot酶类的PR-AUC(%)分布。
可以看到UniZyme几乎在所有家族中保持领先,尤其是中高难度的酶族,曲线整体更平稳。传统方法(DeepDigest、DeepCleave、ProsperousPlus等)在大多数家族的PR-AUC接近零,表明难以跨家族保持良好表现。ReactZyme/ClipZyme的表现比传统模型好,但仍明显低于UniZyme,凸显活性位点先验与统一建模的价值。
案例:HIV-1酶的未见泛化
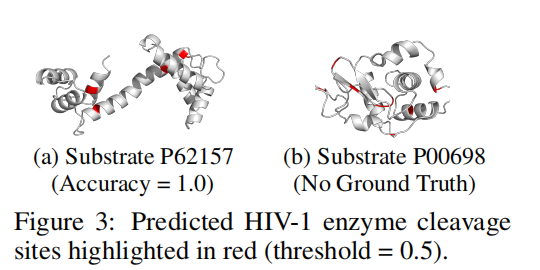
UniZyme在HIV-1酶上的案例预测。
左图(P62157)是一个有已知切割位点的底物,模型在阈值0.5下准确预测了全部4个切割位点(红色高亮),准确率100%。右图(P00698)是一个没有已知标注的底物,UniZyme依然给出了若干高置信度的切割位点预测,为后续实验验证或药物设计提供了候选信息。这一结果说明,UniZyme不仅能在已有标注的情况下表现出色,还能在无标注的新底物中提供位点提示,展现出在新靶点探索和药物研发中的潜力。
总结
UniZyme提出了一种统一的酶切位点预测框架,通过引入生化先验(局部能量挫折、三维距离)与活性位点知识,显著提升了模型在监督设定和零样本设定下的表现。与传统的单酶预测方法相比,UniZyme在跨家族任务上展现了更强的稳健性与泛化能力,并且在HIV-1等案例中证明了其在未见酶和无标注底物上依然能提供可靠预测。
展望
未来,UniZyme及类似方法可以在以下几个方向进一步发展:
结构依赖的缓解:当前模型仍依赖高质量结构预测,未来可融合分子动力学(MD)模拟或构象集合建模,更好捕捉动态结构信息。
跨类型扩展:现阶段聚焦于蛋白水解酶,未来可扩展至更多酶类与不同反应类型,推动更广泛的蛋白功能预测。
机制可解释性:通过进一步结合活性位点解释性分析与实验验证,可提升模型在生物研究与药物设计中的可信度。
临床与药物应用:结合UniZyme的预测能力,可在靶点挖掘、抑制剂设计、抗病毒/抗肿瘤药物开发中提供有力支持,加速AI在药物发现与精准医学领域的落地。
此外,UniZyme已经整合到Protap这一全新蛋白质建模基准中。 Protap覆盖了五大应用场景:其中包括两类专业化任务(蛋白酶催化的切割位点预测、基于PROTAC的靶向蛋白降解)以及三类通用任务(蛋白–配体相互作用预测、蛋白功能预测、突变效应预测)。这一整合不仅验证了UniZyme在酶切预测任务中的优势,也为其在更广泛的下游应用中提供了标准化评测平台。随着Protap基准的不断扩展,UniZyme有望进一步与更多通用预训练模型和领域特定模型进行系统比较,从而推动蛋白质基础模型与应用模型的协同发展。
论文与代码
https://github.com/Ao-LiChen/UniZyme/tree/master
https://arxiv.org/abs/2502.06914
TAILab致力于推动AI+生命科学的融合研究,力求让模型不仅在实验室表现亮眼,更能在实际应用场景中稳定生效。未来,我们将在蛋白质工程、药物设计、功能预测等方向持续深入,探索更多具有可解释性、鲁棒性和跨任务泛化能力的AI方法。
欢迎对蛋白质/酶切/生物AI感兴趣的同学与同行加入我们或交流合作,访问我们的主页了解更多:https://enyandai.github.io/
参考资料
Li, Chenao, Shuo Yan, and Enyan Dai. "UniZyme: A Unified Protein Cleavage Site Predictor Enhanced with Enzyme Active-Site Knowledge." (2025).
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢