单细胞蛋白质组学已广泛应用于当前生物医药研究,但其数据处理长期面临方法选择的困境。一方面,处理流程众多且性能高度地依赖于所研究的具体数据集,增大了对处理流程的选择难度;另一方面,不当的处理易在下游分析中引入偏差,导致生物学解读失真以及研究结论不可重复。

近日,围绕“如何选择最优流程以显著提升处理性能并获得可靠生物学见解”这一单细胞蛋白质组学领域的前沿挑战,浙江大学药学院朱峰教授、付婷婷博士团队在《Nature Protocols》发表研究论文“navigating the data processing for cytometry-based single-cell proteomics”,提出面向单细胞蛋白组学的数据处理工作流优选方法ANPELA(https://idrblab.org/anpela/)。该方法以机器学习与多维度评估为支撑,聚焦细胞亚群鉴定与伪时间轨迹推断两项关键任务,在提升处理稳健性与准确性的同时,为药理机制解析与精准医学研究提供可复用、可推广的单细胞蛋白组学数据处理技术路径。
构建大规模数据处理流程体系
在单细胞蛋白质组学研究中,数以千计的数据处理流程可供研究人员选择。然而,报道指出数据处理流程的性能具有高度的数据依赖性,同一流程在不同数据中表现差异巨大,同一数据在不同处理流程下的分析结果也极不相同,这严重影响了单细胞研究结果的可重复性,导致了相关研究存在严重的“方法选择困难”。此项工作的研究人员以“大规模工作流扫描”和“机器学习评估”为核心,面向单细胞蛋白质组学相关领域的两大关键任务:细胞亚群鉴定(Cell Subpopulation Identification,CSI)与伪时间轨迹推断(Pseudo-time Trajectory Inference,PTI),建立了成体系、可复用的优选策略,帮助研究者根据自有数据推荐最优方法。方法将数据处理拆解为四步:数据补偿(Compensation,消除不同信号通道间的信号溢出),数据转换(Transformation,使数据更接近正态分布),数据归一化(Normalization,减少样本间的非实验因素误差)和信号清洗(Signal Clean,去除数据中的低质量细胞数据)。四大步骤随机组合可产生三千多种工作流,从而为挑选最优数据处理流程奠定了坚实的基础。
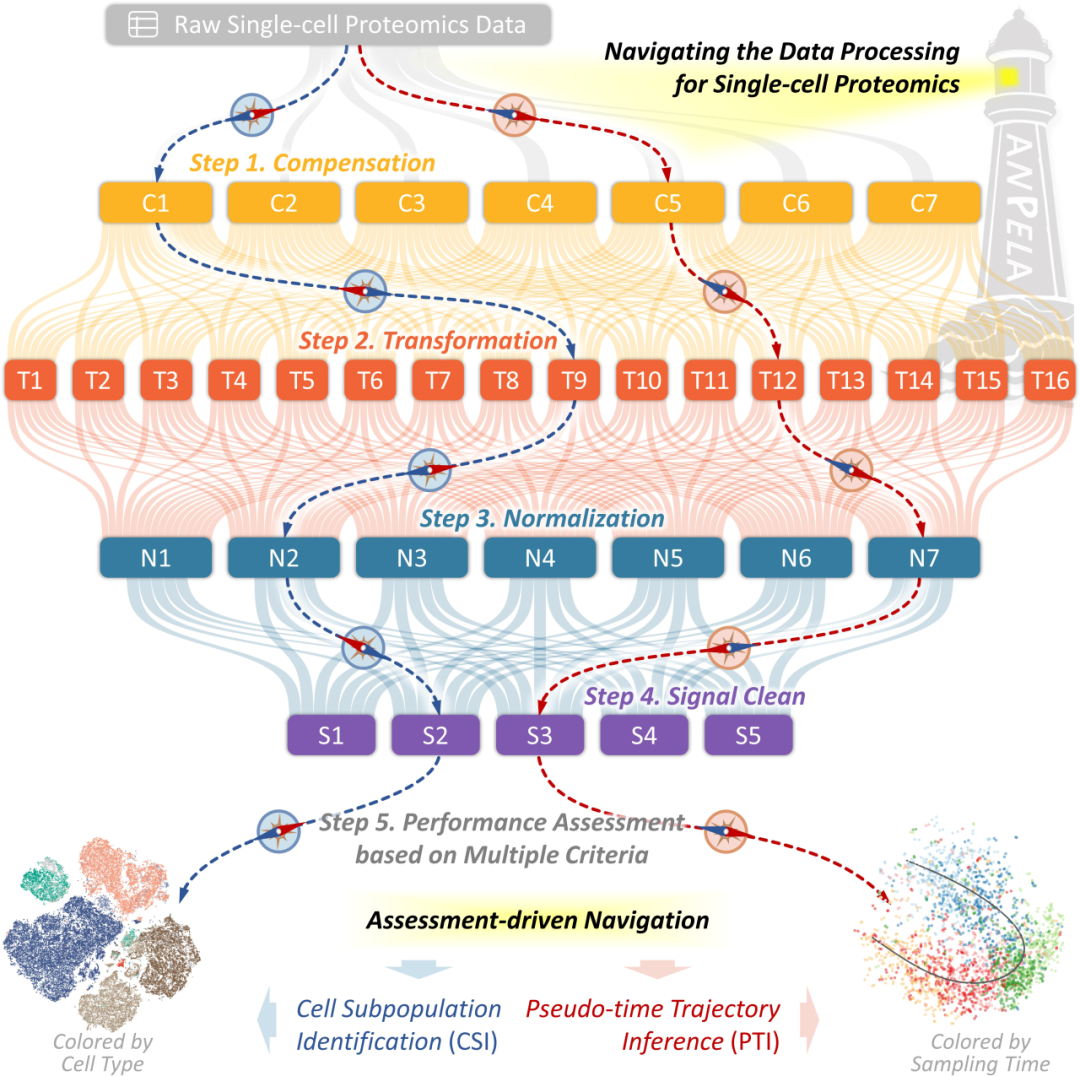
提出多维度综合性能评估策略
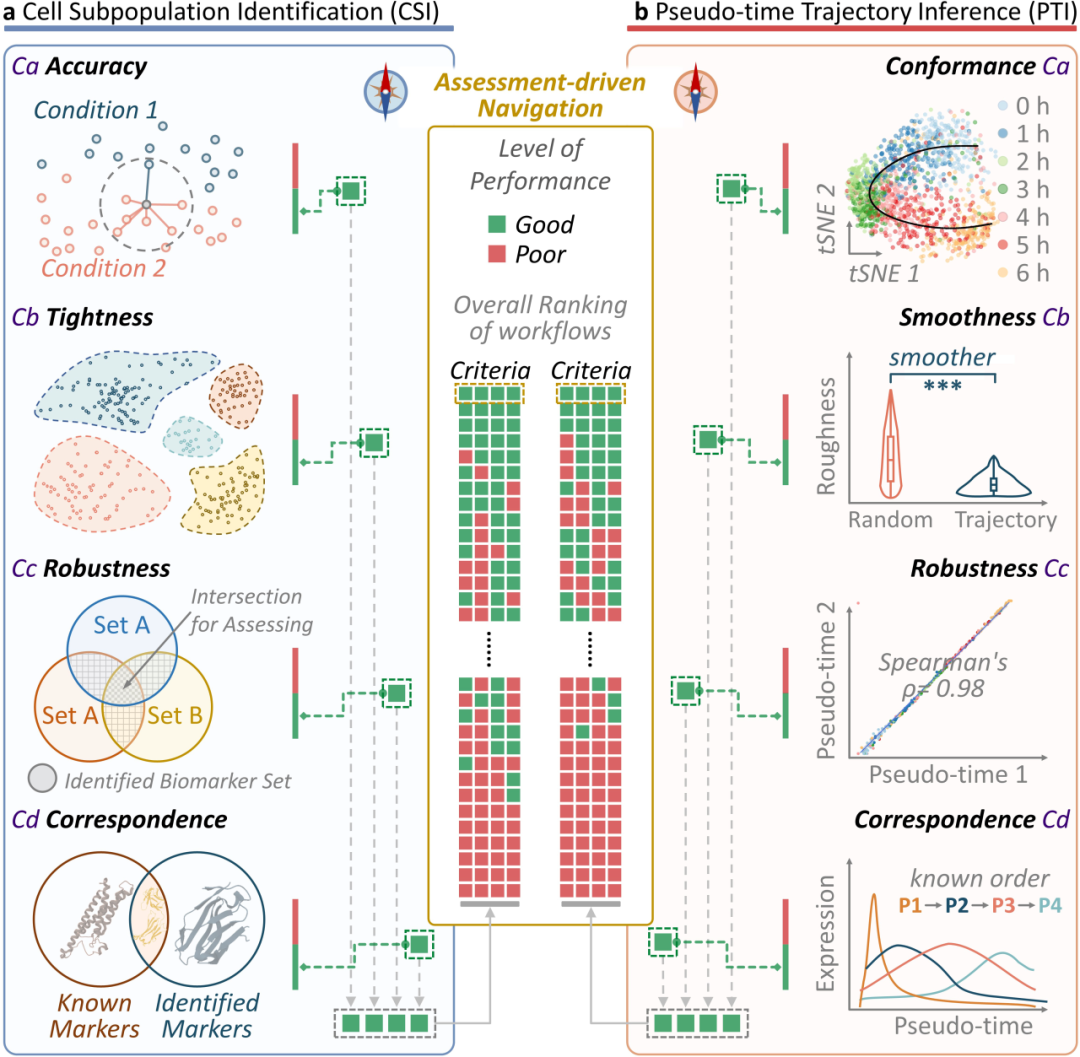
此项工作的研究人员以机器学习为支撑,分别面向CSI与PTI构建了两套成体系、多角度的评估框架。每一框架均由四项相互独立且互补的指标组成,从准确性、稳健性、一致性与先验对应性等维度对候选流程进行整体评价,并以综合评分实现全局排序与自动优选。该评估系统在保证下游结果准确性的同时,显著强化了跨数据集的可重复性与结论稳健性,从方法学层面,为药理学、精准医疗等相关研究提供了确定性与规范化的保障。
保障高准确度的细胞亚群鉴定
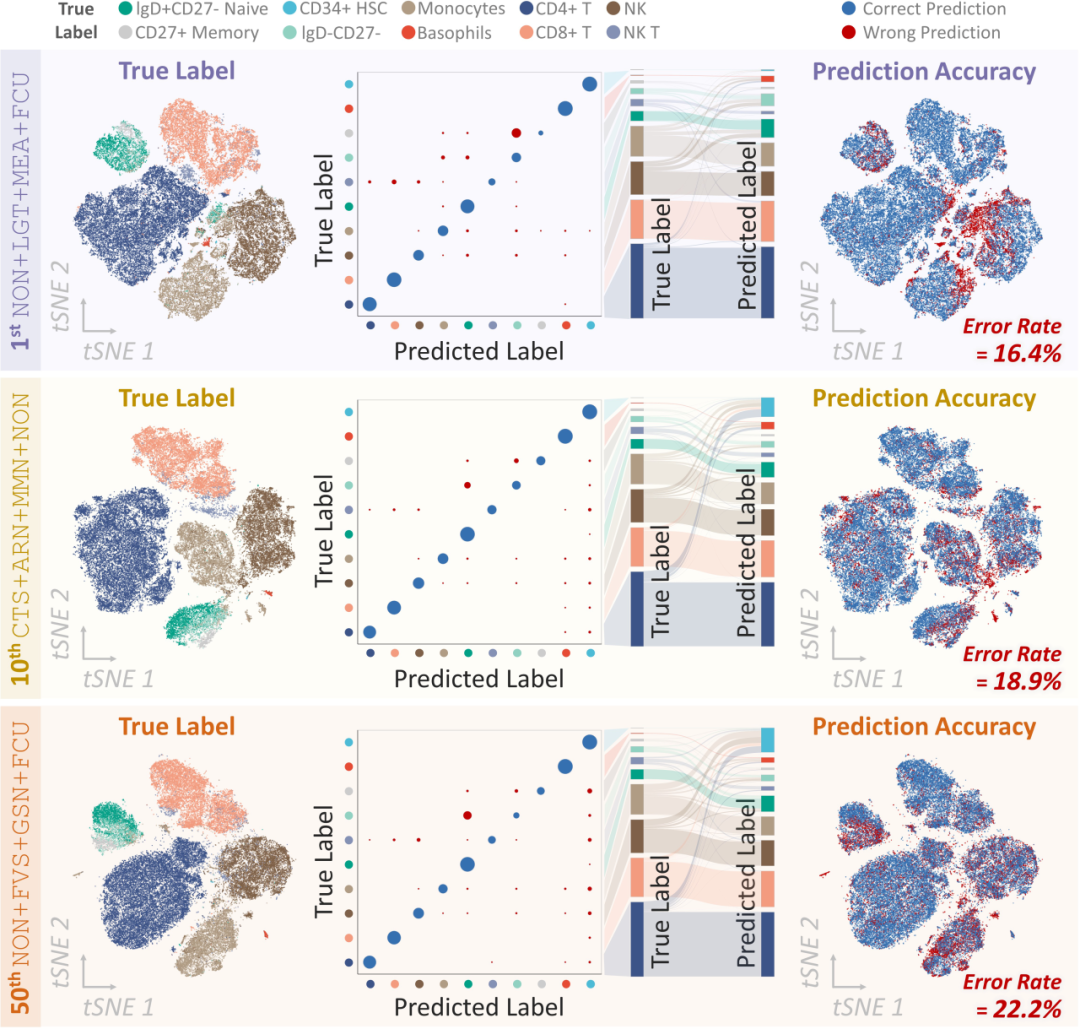
研究人员开展了新冠病毒感染者与健康人群外周血细胞亚群鉴定研究,验证了此项工作所提出方法的效果。针对36例感染者与45名健康人的单细胞蛋白质组学数据,开展了大规模的工作流优选。结果显示,若不加选择地使用领域内常用的处理流程,细胞类型注释的误差可能超出预期(错误率超过20%)。相比之下,研究人员开发的ANPELA可将错误率大幅降低(下降幅度可达6% ~ 20%)。在对CD4⁺ T细胞、CD8⁺ T细胞与嗜碱性粒细胞等关键细胞亚群的鉴定中,ANPELA实现近乎完美的识别(错误率低于1%)。
实现高可信度的细胞轨迹推断
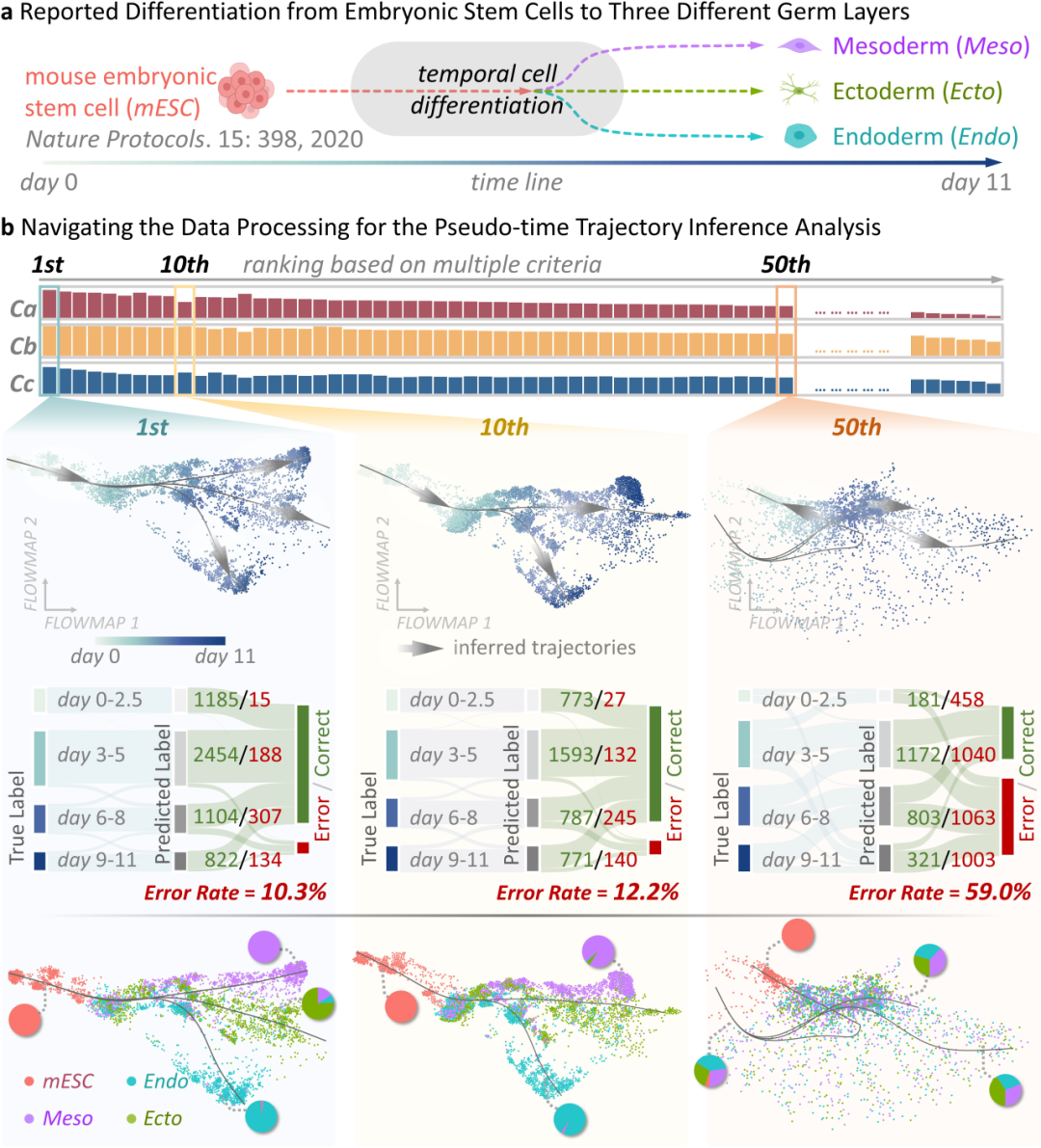
在小鼠胚胎干细胞分化的单细胞蛋白组学数据上,研究团队进一步验证了所提出方法对伪时间轨迹推断的显著增益。该数据覆盖连续11天的多时间点采样,理想的轨迹应自胚胎干细胞出发,沿三条支路分别分化为外胚层、中胚层与内胚层,并在时间序列上与真实生物进程相一致。然而,若直接采用领域内常用的处理流程,分析结果会出现严重偏差,不仅轨迹的起点与终点错误,伪时间与真实时间的误差也超过50%。在部分伪时间与真实时间的误差较低的数据处理结果中,其对应的细胞分化轨迹亦频繁缺失中胚层分支。
相比之下,研究人员开发的ANPELA所推荐的最优流程能够稳定地重建与先验知识高度吻合的三分支发育轨迹,起点与终点分别对应小鼠胚胎干细胞和三类胚层细胞,并在定量评估中实现伪时间与真实时间仅相差10.3%,显著优于领域内常用流程,为疾病发生发展的解析提供了更为可信的细胞分化图景。
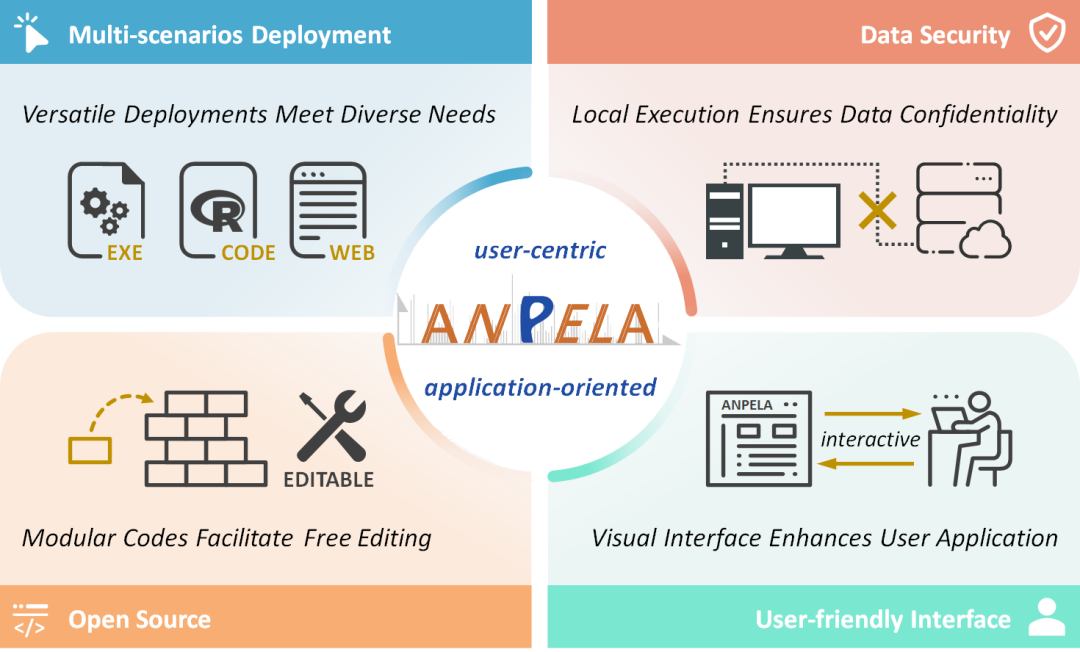
提供多样化的访问和二次开发途径
研究团队在可用性、安全性与可扩展性方面也做了一系列的工作,提供了多样化的访问形式,以保障用户体验。通过桌面软件、R包、在线平台及源代码的多场景部署,降低使用门槛,满足零代码和专业研究者的差异化需求。R包与桌面端均支持本地运行,无需上传私有数据,从源头保障数据安全。全部代码都在GitHub开源,并将功能模块化以确保代码可读性与二次开发友好度。本地与在线版本均配备直观的图形界面,辅以文字教程与交互式教程,帮助用户快速掌握方法要点并顺畅融入既有研究管线。
工具网址:
https://idrblab.org/anpela
参考资料
Sun, H.C., Zhou, Y., Jiang, R.Y., et al. Navigating the data processing for cytometry-based single-cell proteomics. Nature Protoc. (2025)
https://doi.org/10.1038/s41596-025-01257-2
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢