空间组学(Spatial Omics)的最新进展允许研究者对多个组学层(Omics layer)进行空间分辨率测量。然而受限于技术成本与通量,配对空间多组学技术无法得到广泛应用,这导致目前的空间组学数据通常是单一模态的。为了充分利用这些数据,需要开发专门的方法,整合非配对的空间组学数据,以获得新的见解。然而,空间组学数据的特点之一是低信噪比(Signal-To-Noise Ration, SNR),这在转录组以外的模态中尤其明显。如何在噪声背景中识别稳健的生物信号并实现高质量跨组学整合,仍面临实质挑战。
为了解决这一难题,同济大学计算机科学与技术学院陈广教授、同济大学生命科学与技术学院高绍荣院士、张艳平副教授的合作团队开发了名为 SWITCH (Spatially Weighted Multi-omics Integration and Cross-modal Translation with Cycle-mapping Harmonization)的非配对空间多组学数据整合模型。模型借鉴了机器翻译领域中常用的“伪平行语料”思想,在整合过程中动态的生成跨模态翻译作为伪配对,提供额外的对齐信号;同时结合循环一致性损失,确保生成的伪配对的准确性。由此,SWITCH 在无需真实配对数据的条件下,实现多组学数据的稳健对齐与可靠补全,并支撑空间域识别、差异分析、发育轨迹与基因调控网络(GRN)构建等多类下游应用。

方法概述与框架设计
模型的框架主要包含了三个部分:空间感知编码、两阶段对齐和循环映射(图1)。
(1)空间感知表征。针对每一模态,基于空间坐标构建邻近图,引入 GAT 编码器学习空间感知的低维嵌入;再以模态特异的概率解码器回映至原特征空间。
(2)两阶段对齐。先利用知识引导的特征图(guidance graph)开展对抗式初始对齐,得到跨模态的粗对齐表征;随后通过跨模态解码生成“伪配对”,并最小化其与原嵌入的差异,迭代细化对齐质量。
(3)循环映射一致性。通过“模态1→模态2→模态1”的闭环一致性约束,在无真实配对的情形下对翻译结果进行自洽校验,从而提高补全的可用度与稳定性。
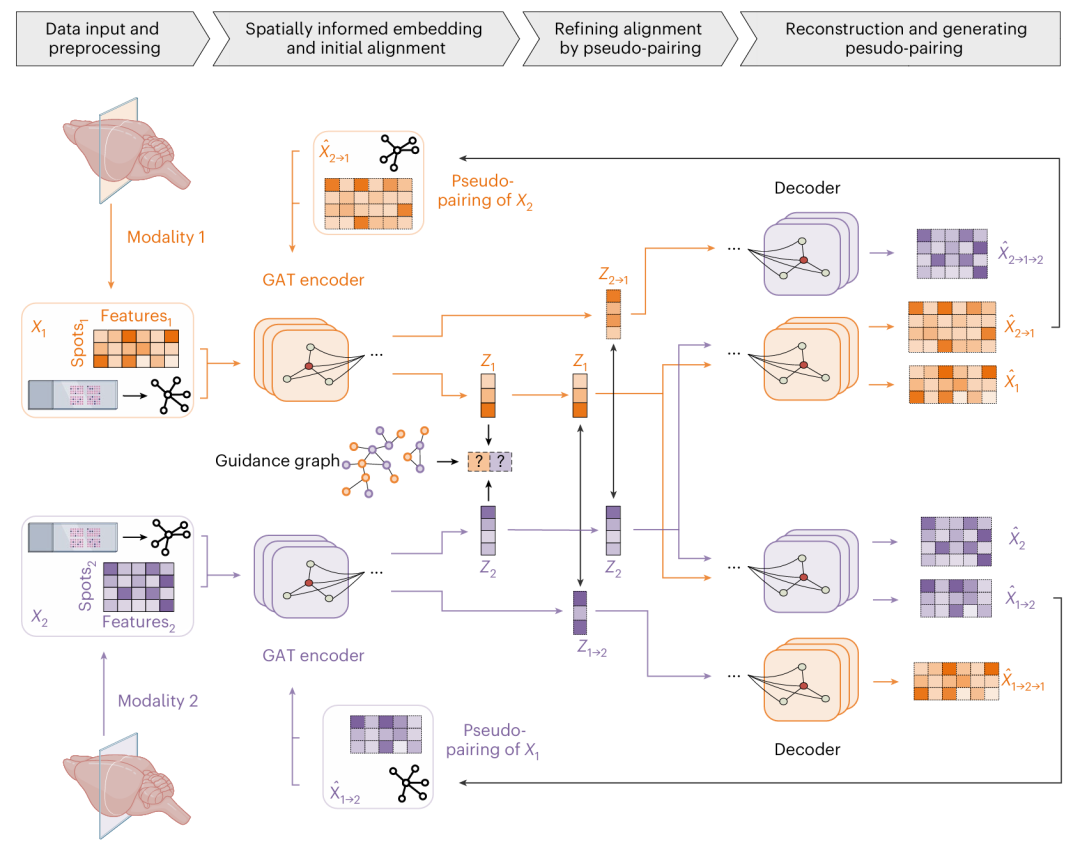
图 1 | 模型的基本框架
基准评测
在具有真实标签的E13小鼠胚胎ATAC–RNA数据中,SWITCH 在组学混合(omics mixing)与生物变异保留(biological conservation)两类目标各 3 项指标的综合评分均优于对比方法,总体得分较次优方法提升 59%。同时在 Moran’s I 与 ICC 指标上取得最佳,显示更高的空间自相关与簇内同质性。基于 spot 层面的对齐评估相较第二名方法,FOSCTTM 降低 46%、FOSKNN 提升约 137%。
在无真实标签、且具备更高分辨率与更低信噪的 小鼠脑 CUT&Tag–RNA 数据中,参考 Allen Brain Atlas 注释,SWITCH 能准确刻画主要解剖区并清晰分层皮层结构(L1/2–3/4/5/6),总体表现优于全部对照方法(图 2)。
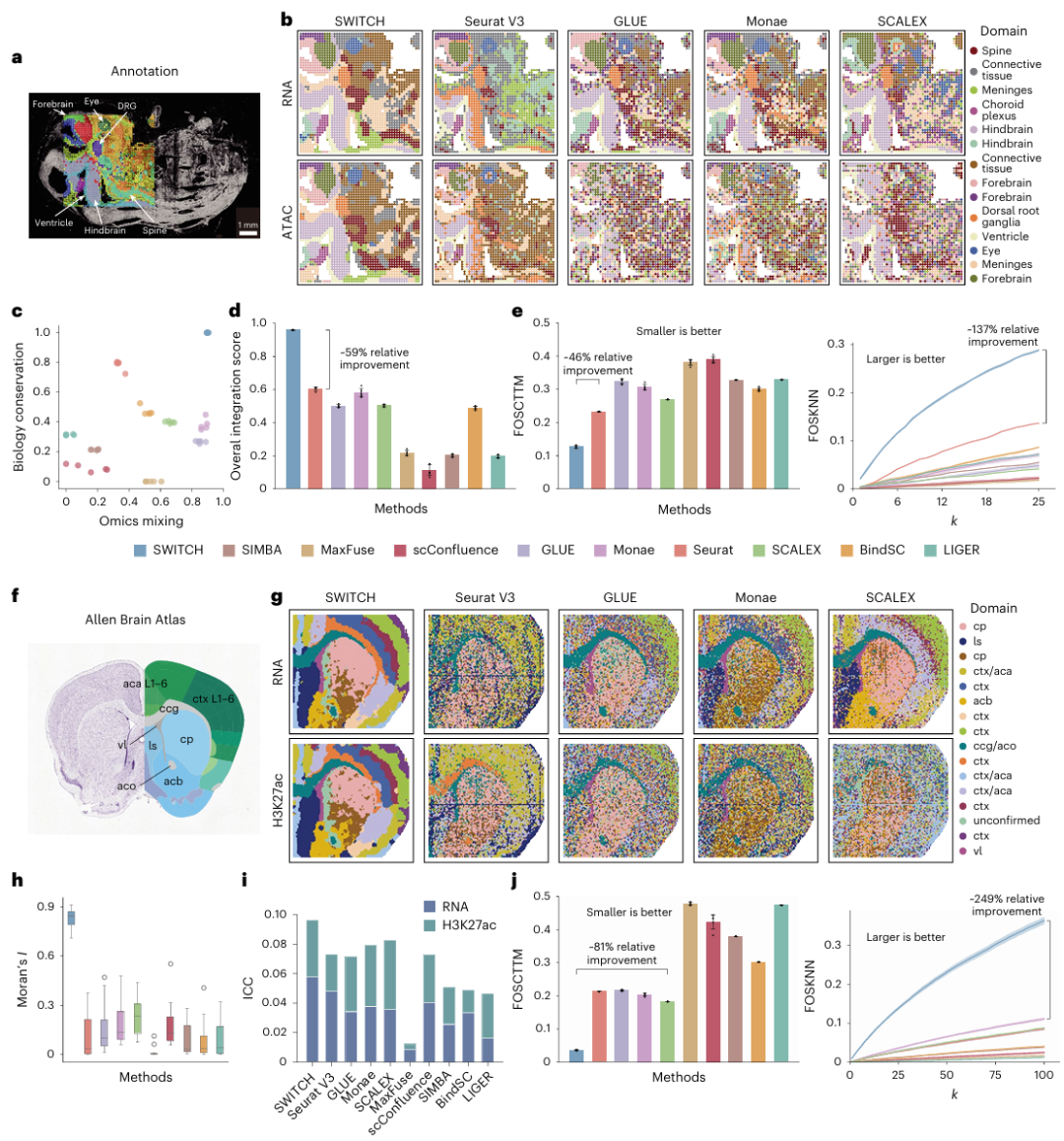
图 2 | SWITCH与其他整合方法的基准评估
跨模态翻译与不确定性估计
在E13小鼠胚胎数据中,SWITCH在整合的同时实现了RNA到ATAC 与 ATAC到RNA 的跨模态插补。受空间数据低敏感度与高噪声影响,插补值与原始观测的 Pearson 相关系数偏低;但当对原始观测进行平滑处理后,插补值与平滑观测之间的相关显著提升,表明模型信号与真实生物变异相一致。同时,SWITCH 的输出包含不确定性估计,且不确定性与误差显著相关,表明其可作为补全质量的内生指标,用于下游分析中的质量控制(图 3)。此外,作者在多种情况下系统评估了SWITCH的跨模态翻译性能,在两套数据的四项翻译任务中,SWITCH 均优于 JAMIE、MultiVI、scButterfly 与 Monae。

图 3 | SWITCH的跨模态插补性能评估
跨模态插补促进下游分析
作者进一步将 SWITCH 应用于整合小鼠胚胎不同发育阶段的多组学数据,识别的空间域与形态学标注高度一致,能够保持清晰的空间域边界,并在 Moran’s I 与 ICC 等无监督指标上获得更优表现(图4)。基于 Monocle3 的皮层发育拟时序分析显示,从脑室带(VZ)到皮层板(CP)的时空梯度在两种模态中一致重现,表明整合的准确性。为了验证插补的准确性,作者使用RNA模态筛选出一系列随拟时序变化的基因,并发现其趋势在ATAC插补的表达中得到保留。此外,代表性的发育相关基因(如 Pax6、Eomes、Tbr1)呈现与既往认知一致的变化模式。进一步地,结合插补后的表达与可及性开展峰–基因关联分析,并与峰–motif 连接信息整合,SWITCH能够构建出具有空间域特异性的基因调控网络(GRN)。
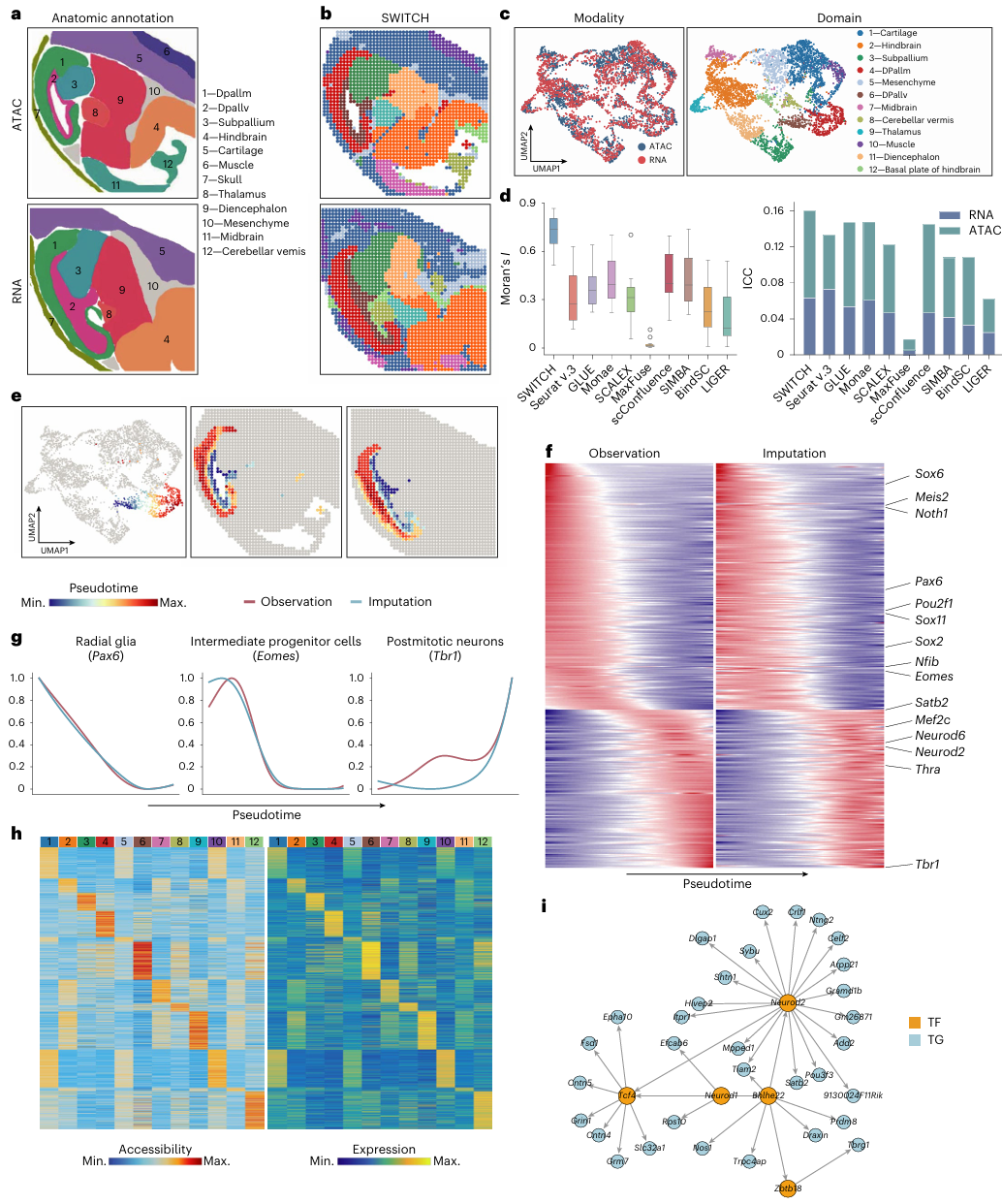
图 4 | SWITCH的跨模态插补支撑的下游分析
单细胞和空间组学的跨模态整合
SWITCH 的模块化设计使其能够很容易的扩展至单细胞与空间组学的跨模态整合。作为示例,作者整合了小鼠大脑的空间转录组(Spatial Transcriptomics, ST)与单细胞 ATAC 数据(图5)。以Allen Brain Atlas为参照,SWITCH在ST切片中能够识别细粒度的解剖结构(如皮层 L1–L6)。在由 ST 向 ATAC 的表达翻译中,作者系统比较了基因表达与其对应转录起始位点(Transcription Start Site, TSS)可及性的空间分布,观察到三类情形:其一,基因呈现明确的空间模式而对应 TSS 不显著;其二,TSS 呈现空间模式而基因不明显;其三,二者在少数情况下具有一致的空间图谱。进一步基于插补的 ATAC 估计转录因子活性分数,可见与相应基因表达具有良好一致性,并与通Allen的原位杂交(In Situ Hybridization, ISH)图像得到验证,表明推断的可及性与调控活性具有较高准确性与生物学可解释性。
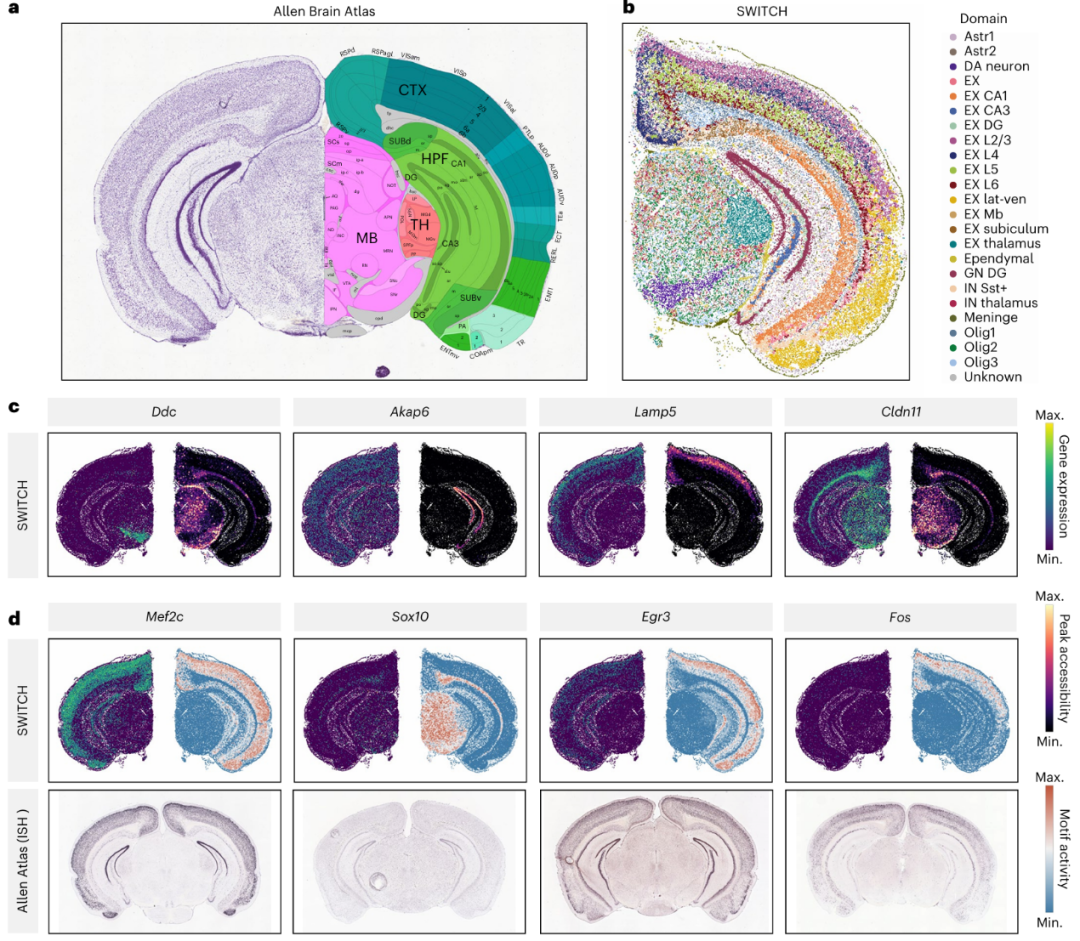
图 5 | SWITCH实现单细胞和空间组学的跨模态整合
参考资料
Li, Z., Qu, S., Liang, H. et al. Integrative deep learning of spatial multi-omics with SWITCH. Nat Comput Sci (2025).
https://doi.org/10.1038/s43588-025-00891-w
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢