三个月后,当研究人员打开小鼠笼子进行最终评估时,对比显而易见:对照组的老年小鼠毛发杂乱、活动迟缓,面对威胁刺激反应迟钝;而接受间歇性哇巴因治疗的同龄小鼠,毛发整洁、姿态警觉,衰弱指数几乎没有恶化。
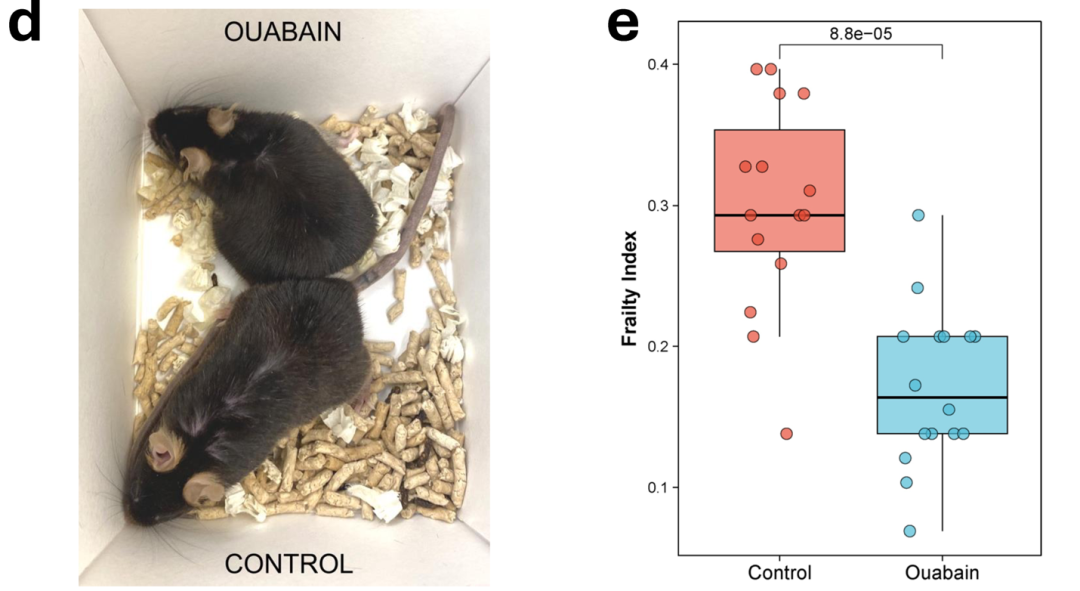
图 1 | 下:对照组老年小鼠,上:接受哇巴因治疗的同龄小鼠。
这个实验验证的,是一个被AI从数万个历史实验中挖掘出的候选物之一。哇巴因,一种临床使用超过百年的心脏糖苷类药物,此前从未被系统性评估过其抗衰老潜力。而识别它的,是一个可以24小时不间断工作的AI智能体,它翻遍了全球科学家在过去二十年积累的海量生物样本数据。
继DNA甲基化时钟和转录组时钟开启生物学年龄精准预测后,如何从海量历史数据中系统性挖掘抗衰老干预措施,成为衰老研究领域的核心挑战之一。面对数百万份样本和数万种可能的干预措施,传统的人工分析在时间和人力上都不可行。

2025年,斯坦福大学的应可钧博士联合哈佛医学院Vadim Gladyshev教授领衔的国际团队发布全球首个自主AI驱动的衰老干预发现平台ClockBase Agent。研究团队还包括来自Genentech、MIT-Harvard Broad研究所、麦吉尔大学等机构的科学家。该平台整合了约200万份人类和小鼠的分子样本数据,应用多种衰老时钟算法,通过AI智能体自主分析数万个干预措施,发现了数千个此前从未被识别的抗衰老候选物。在严格的验证测试中,AI智能体的准确率接近人类专家水平。作为初步验证,研究团队选择了其中一个头部候选物哇巴因进行实验,结果显示其能够显著改善老年小鼠的衰弱指数、心功能和神经炎症。
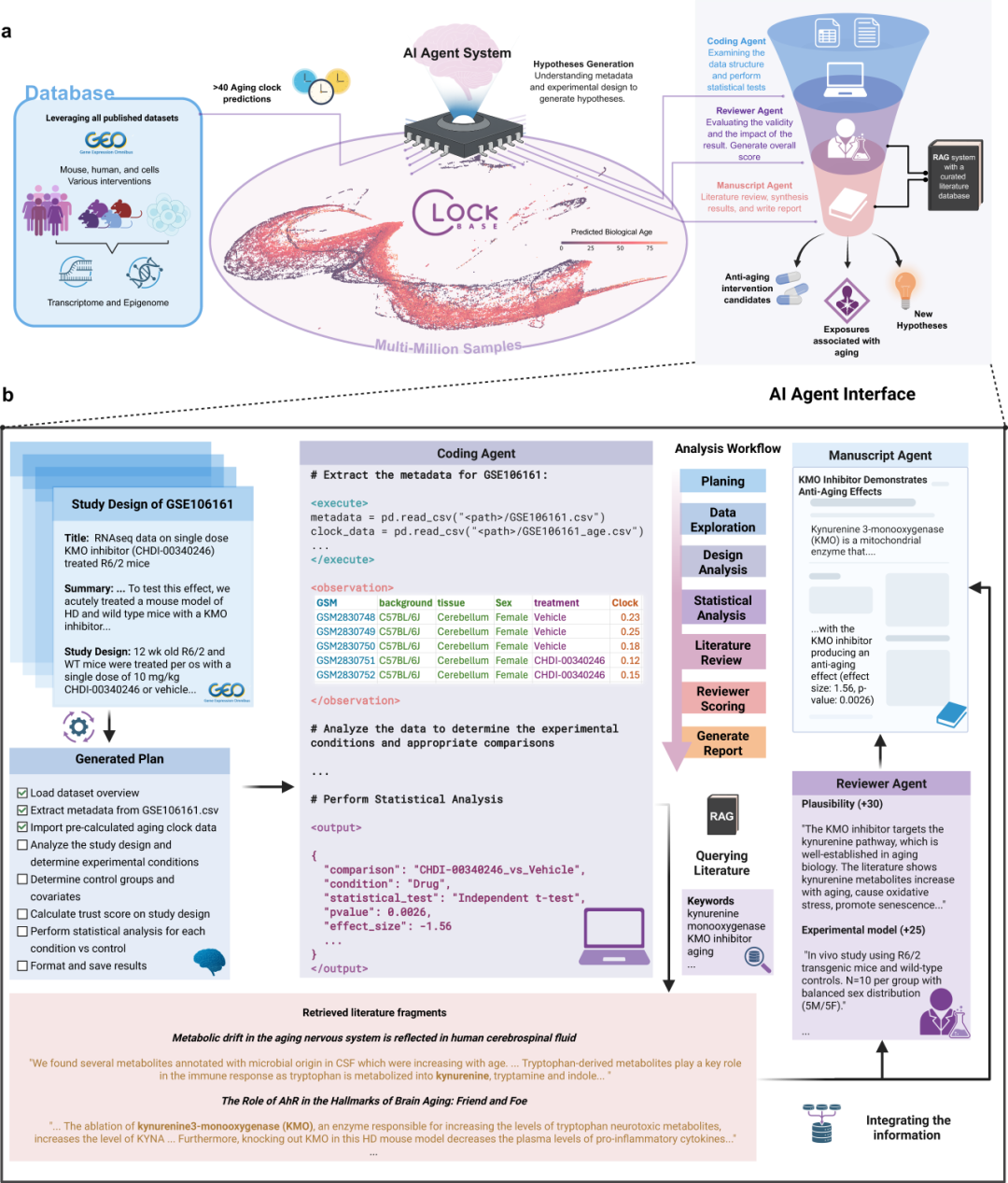
图 2 | ClockBase Agent平台架构与工作流程。a) 系统整合200万份样本、40+衰老时钟和AI智能体框架;b) 以GSE106161为例展示AI智能体的完整分析流程,包括假设生成、统计分析、文献综述和报告撰写。
技术报告:https://www.biorxiv.org/content/10.1101/2023.02.28.530532v4
研究背景:历史数据的系统性重新分析
过去二十年,全球科学家在基因表达数据库(GEO)中沉淀了数百万份人类和小鼠的分子数据。这些数据最初是为了研究癌症、心血管疾病或神经退行性疾病等特定问题而产生。原始研究者测试了数万种药物、基因扰动和环境条件,但几乎从未系统性评估这些干预措施对生物学年龄的影响。
传统研究模式存在系统性局限。单个实验室通常只分析自己的数据集,缺乏跨研究的整合视角。即使有研究者意识到重新分析的价值,应用多种衰老时钟、处理异构数据和执行标准化统计分析都需要专业的生物信息学背景。面对数万个研究,人工逐一分析在时间和人力上不可行。
ClockBase Agent系统性解决了这一问题。平台整合了来自GEO数据库的超过200万份样本,包括人类和小鼠的DNA甲基化与RNA测序数据,覆盖数千种组织类型和实验条件。
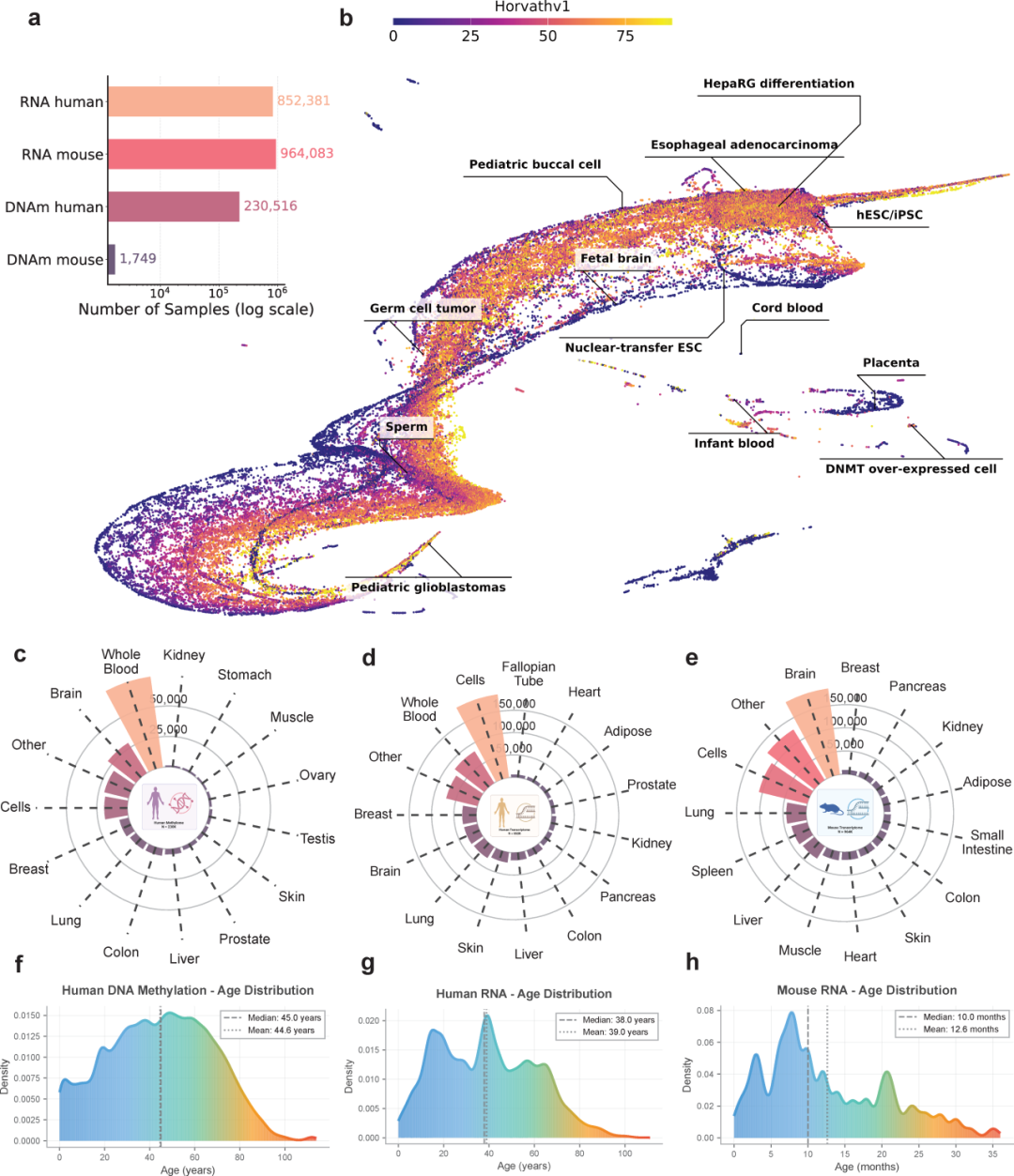
图 3 | 200万样本的综合生物学年龄图谱。a) 四类数据的样本分布;b) 人类DNA甲基化样本的UMAP可视化,颜色代表Horvath时钟预测的生物学年龄;c-e) 人类DNA甲基化、人类RNA和小鼠RNA的组织分布;f-h) 三类数据的年龄分布密度图。
方法学创新:多智能体协同分析系统
ClockBase Agent的核心创新在于构建了一个完全自主的多智能体系统。系统由三个专门化的智能体模块组成:分析执行智能体负责解析数据库中的元数据,识别实验设计并自主选择合适的统计方法;生物学解释智能体检索大量近期衰老研究文献,将统计发现转化为生物学意义和机制假说;评分智能体从生物学合理性、实验模型质量、转化潜力和机制新颖性等多个维度对每个候选物进行综合评分。
为确保分析质量,研究团队建立了严格的验证流程。两位独立的博士级生物信息学家对随机选择的分析结果进行了盲法评估,从假设生成、统计检验选择到效应量估计等多个关键维度进行审核。结果显示,AI智能体的准确率超过99%,接近人类专家水平。
系统的评分框架综合考虑统计显著性和生物学转化价值。评分算法整合了生物学合理性、实验模型质量、衰老通路相关性、实验严谨性、转化潜力以及机制新颖性等多个维度。这套评分体系旨在优先识别那些机制清晰、实验严谨且具有临床转化前景的候选物。
分析结果:数万个干预措施的系统性评估
系统重新分析了上万个小鼠RNA-seq研究,涵盖数万个干预-对照比较。分析范围包括基因扰动、药物治疗、环境暴露和疾病模型。统计分析显示,约13%的干预措施显著影响生物学年龄,其中数百个干预获得了很高的综合评分。
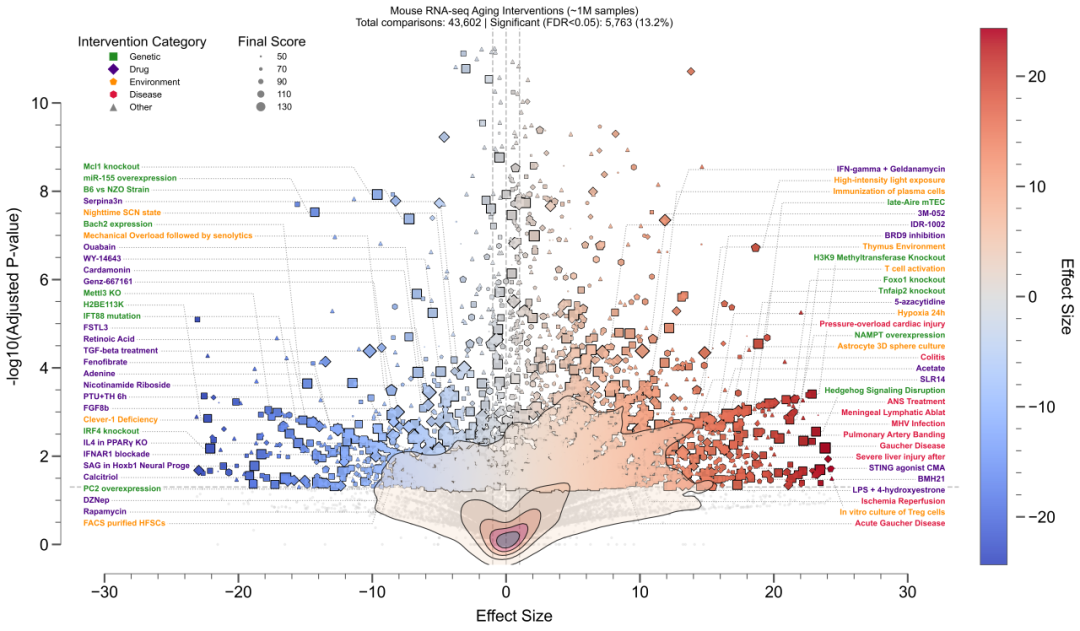
图 4 | AI智能体识别的数万个干预的全景图。火山图展示了不同类别干预(基因、药物、环境、疾病)对生物学年龄的影响,点的大小代表综合评分。
在药物干预类别中,系统识别出数百个显著影响转录组年龄的化合物。综合评分较高的候选物包括哇巴因(一种具有衰老细胞清除活性的心脏糖苷)、经典的mTOR抑制剂雷帕霉素、以及临床常用的降脂药非诺贝特。在基因扰动类别中,IRF4敲除(免疫细胞分化关键因子)和Mettl3敲除(RNA甲基化调控)也获得了很高的评分。环境因素分析显示,机械负荷结合衰老细胞清除治疗获得了最高评分,而高强度光照和缺氧环境则显著加速衰老进程。
为验证发现的可靠性,研究团队将所有高置信度候选物与已发表的长寿数据库进行了交叉比对。部分基因干预与GenAge数据库重叠,包括Grn、Pten、Neil1和Apoe等已知长寿基因,方向一致性超过80%。部分药物也与DrugAge数据库重叠,涵盖雷帕霉素、烟酰胺核糖、槲皮素等经典抗衰老化合物。值得注意的是,超过97%的候选物是数据库中未记录的新发现,提示系统性重新分析可能识别出大量此前未被关注的干预措施。
实验验证案例:以哇巴因为例
为验证系统识别的候选物的有效性,研究团队选择了综合评分最高的化合物之一哇巴因进行概念验证实验。哇巴因是一种心脏糖苷类化合物,已知具有衰老细胞清除活性,但此前尚未有研究系统性评估其对整体衰老表型的影响。这只是数千个候选物中的一个验证案例,平台识别的其他候选物也有待进一步研究。
研究团队设计了一个为期三个月的间歇性给药实验,老年雄性小鼠接受每周两次的哇巴因治疗。实验结束时,研究人员从转录组年龄、衰弱指数、心脏功能、代谢和神经炎症等多个维度评估了治疗效果。
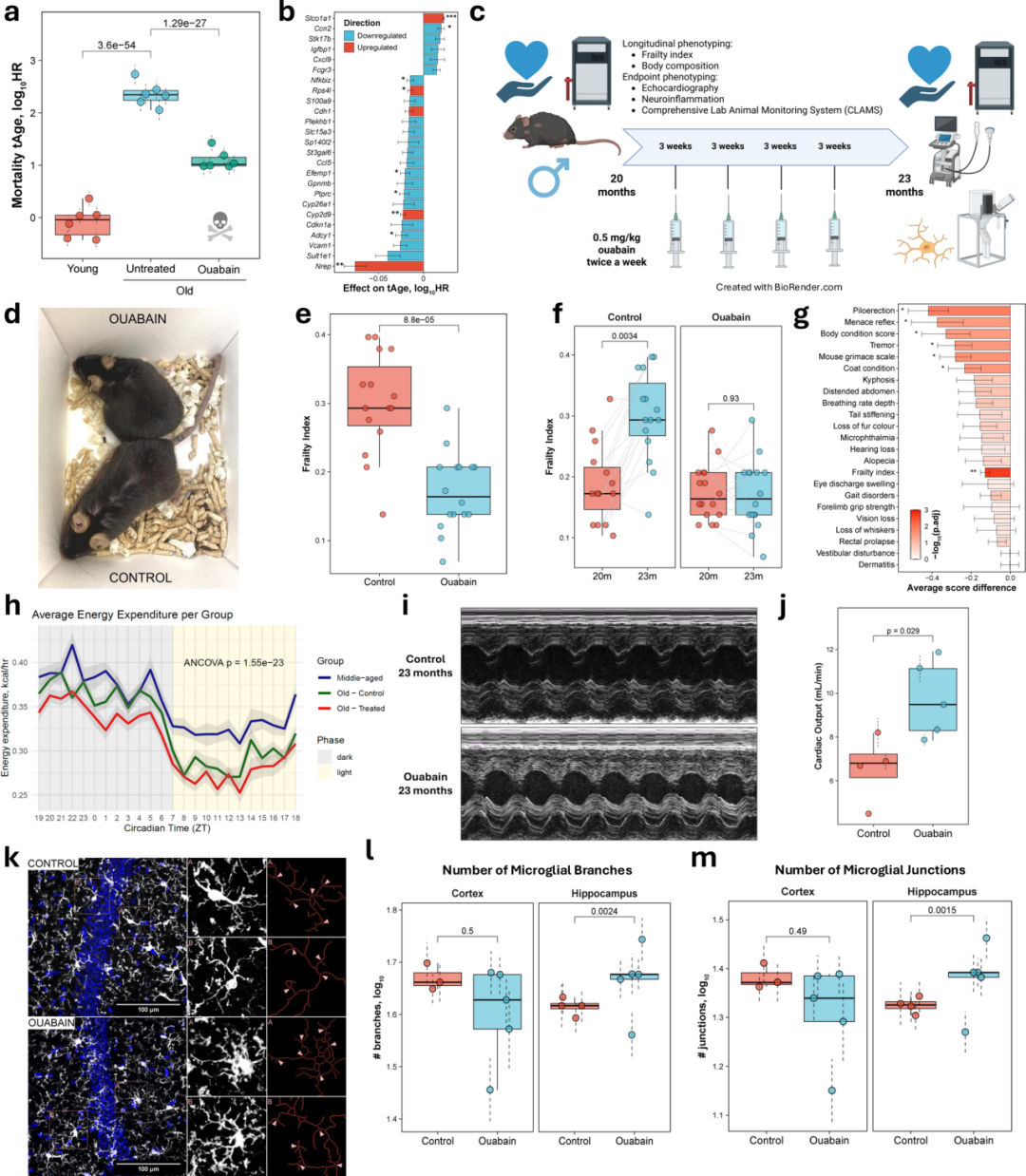
图 5 | 哇巴因的系统性抗衰老效应验证。a) 转录组年龄显著降低;b) 关键贡献基因(Nrep上调);c) 实验时间线;d-g) 衰弱指数完全阻止进展;h) 能量代谢的适度调整;i-j) 心输出量显著提高;k-m) 神经炎症降低(小胶质细胞分支和连接点增加)。
根据小鼠多组织死亡率预测时钟,哇巴因治疗显著降低了老年小鼠的转录组年龄。基因层面的分析揭示,Nrep(神经元再生相关蛋白)的表达显著上调,这一基因此前被报道在异体共生(heterochronic parabiosis)实验中也显著上调,提示哇巴因可能通过相似的再生机制发挥作用。模块特异性时钟进一步显示,炎症和干扰素信号通路被下调,而Nrf2抗氧化通路、氧化磷酸化以及mRNA剪接和翻译等关键过程得到优化。
衰弱指数分析显示,对照组小鼠的衰弱指数在实验期间显著上升,而哇巴因治疗组的衰弱指数保持相对稳定。治疗组小鼠在毛发、反射、身体状况等多个衰弱参数上显著优于对照组,未观察到治疗相关的不良反应。
心脏功能分析显示,超声心动图检测中哇巴因治疗组的心输出量明显高于对照组,符合心脏糖苷类药物增强心肌收缩力的经典药理机制。
神经系统分析发现,哇巴因治疗组海马区的小胶质细胞形态更接近健康的巡视状态而非激活状态,提示神经炎症水平降低。这与神经功能改善相一致。
代谢层面,哇巴因治疗产生了能量消耗的适度调整,这可能反映了代谢效率的优化。重要的是,这种代谢调整并未伴随食物摄入或活动量的改变,提示其为内在代谢重编程的结果。
数据与工具的开放共享
ClockBase Agent平台已上线(www.clockbase.org),向科研界开放所有数据和分析工具。
潜在应用场景包括:衰老靶标发现、药物再利用筛选、分子机制研究,以及跨物种验证研究。研究团队持续收集用户反馈,以改进系统性能和分析准确性。
研究展望
ClockBase Agent展示了AI辅助系统性数据重新分析的可行性。这一方法提供了几个潜在优势:AI可以持续扫描所有数据,识别人类研究者可能忽略的模式;系统性整合历史数据,提高已投入资源的利用效率;降低生物标志物分析的技术门槛;随着新数据发布和新算法开发自动更新分析结果。
哇巴因的成功验证只是一个开始。平台识别的数千个候选物中,许多具有不同的作用机制和转化潜力,有待系统性的实验验证。研究团队正在进行多个方向的扩展。规模层面,团队正在处理更多人类样本数据。验证层面,将纳入人类队列研究数据,评估小鼠发现在人类中的可重复性。此外,该AI智能体框架可以扩展到癌症、免疫、代谢等其他生物学问题。
致谢:感谢Biomarkers of Aging Consortium成员的宝贵反馈,感谢Biolearn团队提供衰老生物标志物标准化工具,感谢OpenAI团队和Avinasi Labs提供API支持。
参考资料
Autonomous AI Agents Discover Aging Interventions from Millions of Molecular Profiles. Kejun Ying, Alexander Tyshkovskiy, Alibek Moldakozhayev, Hanchen Wang, Cecília G. De Magalhães, Sharif Iqbal, Amanda E. Garza, Albina Tskhay, Jesse R. Poganik, Kexin Huang, Yuanhao Qu, Dmitrii Glubokov, Cheng Jin, Donghyun Lee, Hanna Liu, Carolina Leote, Alexandre Trapp, Lucas Paulo de Lima Camillo, Csaba Kerepesi, Mahdi Moqri, Odin Zhang, Kaiyi Jiang, Fedor Galkin, Alex Zhavoronkov, Jeremy M. Van Raamsdonk, Mengdi Wang, Le Cong, Aviv Regev, Jure Leskovec, Tony Wyss-Coray, Vadim N. Gladyshev; bioRxiv 2023.02.28.530532
doi: https://doi.org/10.1101/2023.02.28.530532
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢