【论文标题】Improving Biomedical Pretrained Language Models with Knowledge
【作者团队】Zheng Yuan, Yijia Liu, Chuanqi Tan, Songfang Huang, Fei Huang
【发表时间】2021/04/19
【机 构】清华 & 阿里巴巴,美国
【论文链接】https://arxiv.org/pdf/2104.10344v1.pdf
【代码链接】https://github.com/GanjinZero/KeBioLM
【推荐理由】生物医学NLP预训练的又一进步
预训练语言模型已经在许多自然语言处理任务中表现优秀,而许多工作正在探索将知识纳入语言模型。在生物医学领域,专家们花了几十年的精力来建立大规模的知识库,例如,统一医学语言系统(UMLS)包含数以百万计的实体及其同义词,并定义了实体之间的数百种关系。利用这些知识可以使各种下游任务受益,如命名实体识别和关系提取。为此,本文提出了KeBioLM,一个生物医学的预训练语言模型,明确地利用UMLS知识库中的知识。具体来说,作者从PubMed的摘要中提取实体,并将它们与UMLS联系起来。然后,作者训练了一个知识感知的语言模型,应用纯文本编码层来学习实体表示,同时应用文本-实体融合编码来聚合实体表示。此外,作者增加了两个训练目标,即实体检测和实体链接,对BLURB基准的命名实体识别和关系提取的实验证明了本文方法的有效性。对收集到的探测数据集的进一步分析表明,本文的模型有更好的能力来模拟医学知识。
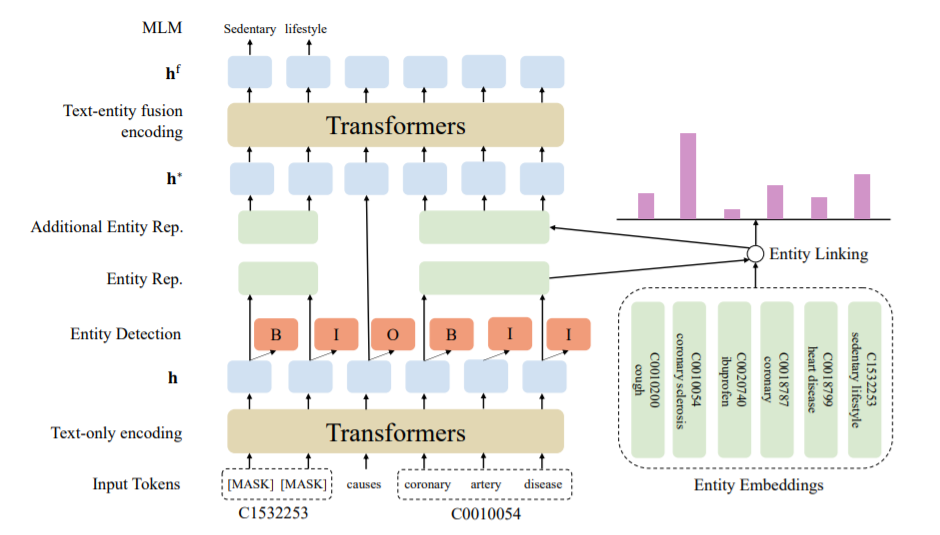
上图为KeBioLM的框架。本文在MLM以外加入了其他2个预训练任务,实体探测(对每个实体进行BIO编码,监督式任务)和实体链接(实体和其他实体相似度),将单纯的文本通过纯文本编码层编码学习文本表征,通过预训练任务学习实体表征,随后应用文本-实体融合编码来聚合实体表征和文本表征。
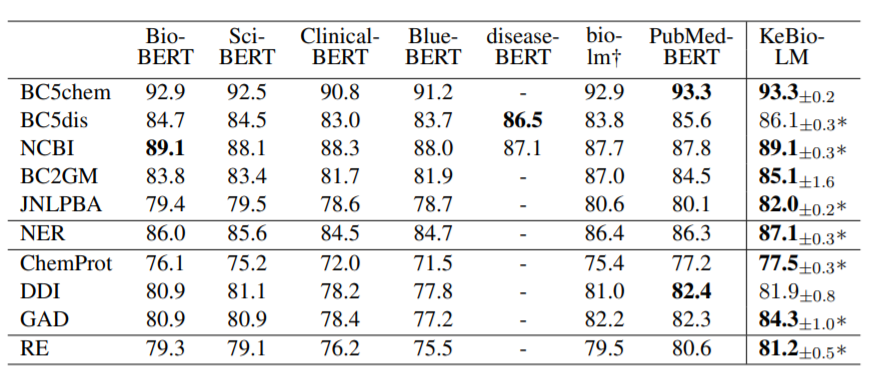
上图显示了在BLURB基准中,NER(实体识别)和RE(关系抽取)任务的F1分数,KeBioLM的标准偏差运行5次中报告的。diseaseBERT-biobert和bio-lm的结果来自其相应的论文,其他的是源自BLURB。*表示单样本t检验的p ≤ 0.05,Bio-lm采用了与BLURB不同的衡量标准(微观F1与宏观F1),因此,这里只是列出它的结果,但不直接与之比较。
比较而言KeBioLM的平均性能优于PubMedBERT并总体表现优异。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢