深度学习的潜力在蛋白质结构预测界已经被认识了一段时间,在CASP13之后变得无可争议。在CASP14中,深度学习将该领域提升到了意料之外的水平,达到了接近实验的准确性。这一成功来自于从其他机器学习领域迁移过来的进展,以及专门设计来处理蛋白质序列和结构的方法。新出现的方法包括:(i) 几何学习,即在图、三维Voronoi镶嵌和点云等表征上学习;(ii) 利用注意力的预训练蛋白质语言模型;(iii) 保留三维空间对称性的等价架构;(iv) 使用大型元基因组数据库;(v) 蛋白质表征的组合;(vi) 最后是真正的端到端架构,即从序列开始并返回三维结构的可区分模型。作者对过去两年中开发的并在CASP14中广泛使用的新型深度学习方法进行了概述并提出了自己的看法。
上图为CASP14中基于深度学习的方法的输入和输出的示意图,不包括将几个不同来源的方法集成成的pipline方法,以及缺乏明确描述的方法。蓝线和红线分别表示输入和输出水平。
- 预训练:由在大量序列数据上预训练的NLP模型决定的序列embedding。
- MSA:原始多重序列比对。
- MSA特征:如PSSMs,协方差和精度矩阵。
- 连接图:连接或距离矩阵。
- 几何:几何特征,通常包括包括接触/距离和扭转角。
- 结构:三维坐标。
- QA: 模型质量。

上图为预训练序列模型在Casp14中的应用。我们可以在数以亿计的自然序列上获得的知识,以产生特定的表征embedding。NLP一些经典模型包括BERT、ELMo。 和GPT-2已被调整为 "蛋白质语言"。在半监督的预训练阶段,模型试图预测一个被掩盖的或下一个标记。 在CASP14中,EMBER直接利用了ELMo 和BERT,而HMS-Casper则使用了后者的一个重新整理的版本,称为AminoBert。A2I2Prot和 CUTSP利用了TAPE预训练模型。
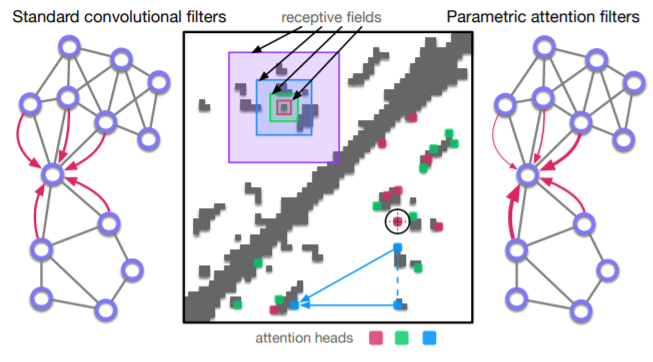
上图为蛋白卷积滤波器和注意力Head之间的比较。卷积滤波器和注意力头之间的比较。输入的数据可以表示为二维图像也可为图。编码的信息可以是MSA推断的协方差或模板衍生的蛋白质残基间的欧氏距离。在图像的左上角三角形中,重叠的方块对应于通过在2DCNN中堆叠多层卷积滤波器而获得的不断增加的感受野。在主对角线下的区域,彩色的方块代表输入点相对于圆心来说是最重要的,这是应用注意力滤波器的结果。如果节点代表残基,那么注意力的权重可以用三维距离或接触来解释。 在左边的图中,红色箭头表示一个 标准的卷积,聚集了来自 相邻节点的信息,而在右边,注意力机制将更多的权重放在某些邻居身上 (用箭头的粗细来说明)。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢