近年来,在生物医学领域利用图神经网络解决药物相关的问题飞速发展。然而,像其他任何深度学习架构一样,GNNs也是数据敏感的。虽然在现实世界中需要标签往往是昂贵的,但以无监督的方式对GNN进行预训练已被积极探索。其中,图对比学习,通过最大化成对的图增强之间的相互信息,已被证明对各种下游任务有效。然而,目前的图对比学习框架有两个限制。首先,数据增强是为一般的图设计的,因此对某些领域可能不适合或不够强大。第二,对比方案只学习对局部扰动不变的表征并没有考虑数据集的全局结构,而全局特征对于下游任务也可能是有用的。因此,本文提出了一个生物医学领域中的分子图对比学习新的框架,MoCL,一种利用局部和全局层面的领域知识来帮助表示学习的方法。局部层面的领域知识指导着增强过程,从而在不改变图形语义的情况下引入变化。全局层面的知识对整个数据集中的图之间的相似性信息进行编码,帮助学习具有更丰富语义的表示。整个模型是通过一个双重对比目标来学习的。作者在各种分子数据集上评估了MoCL的线性和半监督设置,结果显示MoCL达到了最先进的性能。
- 作者提出了一种新的基于局部领域知识的分子图的增强方案,使图的语义在增强过程中不发生变化。
- 作者建议通过增加一个利用分子图之间相似性信息的全局对比损失,将数据的全局结构编码到图的表示中。
- 作者提供的理论依据是,学习目标与度量学习中的三重损失有关。阐明了整个框架的有效性。
- 作者在各种分子数据集上对MoCL进行了评估,包括线性和半监督学习。在线性和半监督设置下,对MoCL进行了评估,并证明了它比最先进的方法更有优势。
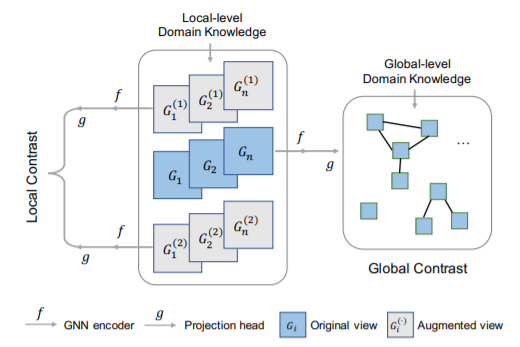
上为MoCL的整体框架。两个增强的视图是由局部层面的领域知识生成的。然后,与原始视图(蓝色)一起被送入GNN编码器和投影的头。局部水平的对比学习使两个增强的视图之间的MI最大化,全局层面的对比学习最大化了两个相似图之间的MI。
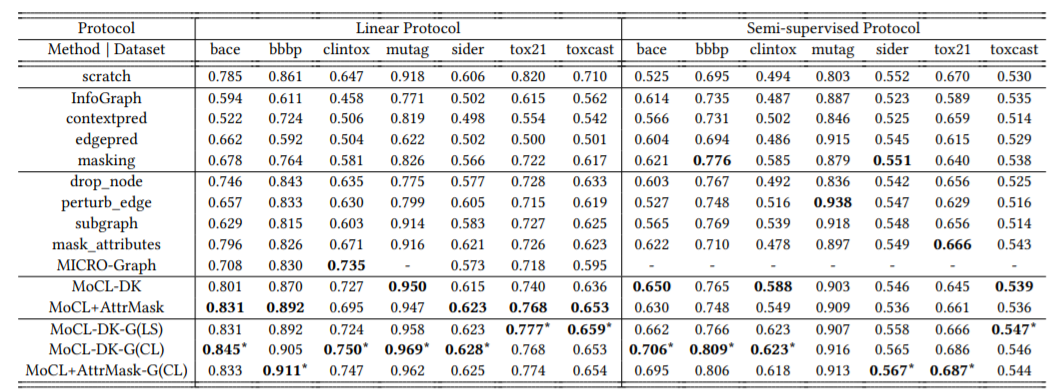
线性和半监督protocol下比较方法的平均测试AUC(5次运行)
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢