【论文标题】SPOT-Contact-Single: Improving Single-Sequence-Based Prediction of Protein Contact Map using a Transformer Language Model, Large Training Set and Ensembled Deep Learning
【作者团队】Jaspreet Singh, Thomas Litfin, Jaswinder Singh, Kuldip Paliwal, Yaoqi Zhou
【发表时间】2021/06/20
【机 构】深圳湾实验室,格里菲斯大学
【论文链接】https://www.biorxiv.org/content/10.1101/2021.06.19.449089v1
准确预测蛋白质接触图对于准确预测蛋白质结构和功能至关重要。因此,许多方法已经被开发出来用于蛋白质接触图的预测。然而,大多数接触图的预测方法依赖于蛋白质序列的进化信息,许多蛋白质由于缺乏序列同源性,可能不存在这种信息。此外,生成进化图谱需要大量的计算和时间。因此,我们开发了一个接触图预测器,利用预先训练好的语言模型ESM-1B的输出作为输入,再加上一个大的训练集和一个残差神经网络的集成。我们表明,所提出的方法比基于单序列的预测器SSCpred有明显的改进,在独立的CASP14-FM测试集上,F1分数提高了15%。它也优于基于进化谱系的方法TrRosetta和SPOT-Contact,在SPOT-2018的无同源蛋白集(Neff=1)上的F1得分分别提高了48.7%和48.5%。新方法提供了一个比基于profile的方法更快、更准确的替代方法,特别是对大规模预测非常有用。
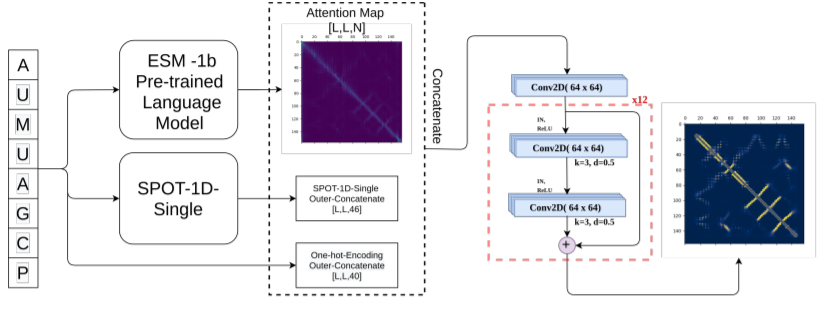
上图显示了模型流程,我们研究了使用ESM-1b的注意力图作为我们模型的输入特征,以改善我们基于单序列的方法的接触图预测。我们证明了将无监督学习特征与one hot编码和SPOT-1D-Single的输出连接起来的结果在所有的蛋白质上都优于基于单序列的SSCpred,在同源蛋白质有效数量较少的情况下,优于基于MSA的预测器。我们还表明,通过不同的训练方法和不同的特征组合训练出来的模型的集合也会增加这种改进。

上图显示了模型集成的策略
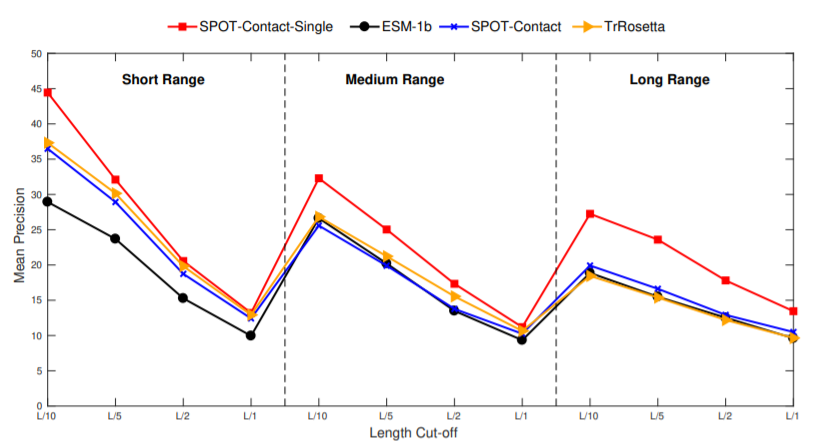
上图显示了各个方法分别在短程,中程,长程上预测的准确度,可以看的本文方法在长程表现优秀。单纯的预训练模型效果总体上最差。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢