识别药物-蛋白质相互作用(DPI)在药物发现中是至关重要的,目前已经开发了许多机器学习方法来预测DPI。现有的方法通常使用不真实的数据集和隐藏的偏见,这将限制虚拟筛选方法的准确性。同时,大多数DPI预测方法更关注分子表征,缺乏对蛋白质表征和不同实例之间高层次关联的有效研究。为此,我们在此提出了一个新的结构感知的多模式DPI预测模型--X-DPI,该模型在一个精心策划的工业规模的基准数据集上执行。
我们为DPI预测建立了一个高质量的基准数据集,名为GalaxyDB。这个工业规模的数据集与无偏的训练过程一起,形成了一个更强大的基准研究。为了获得蛋白质的信息,我们通过结合预测的接触图和图神经网络,从蛋白质序列中构建了一个结构感知的图神经网络方法。通过进一步整合基于结构的表征和针对分子和蛋白质的高水平预训练嵌入,我们的模型更有效地捕获了它们之间相互作用的特征表征。结果,X-DPI超过了最先进的DPI预测方法,在DAVIS数据集中获得了5.30%的均方误差(MSE),在GalaxyDB数据集中获得了8.89%的曲线下面积(AUC)。此外,我们的模型是一个可解释的模型,具有基于transformer的相互作用机制,可以准确地揭示分子和蛋白质之间的结合点。
本文重点:
- 我们整理了一个名为GalaxyDB的高质量基准数据集,专门用于基于机器学习的虚拟筛选。GalaxyDB来源于ExcapeDB,由372个常见的 靶点,有381,021个确认的活性化合物和1,634,038个确认的非活性化合物。大规模的数据集和无偏的训练程序为模型的建立提供基础。
- 为了获得丰富的蛋白质信息,我们将蛋白质序列表示为蛋白质图。我们通过结合预测的接触图将蛋白质序列表示为一个蛋白质图。同时,我们从蛋白质序列中构建了一个结构感知的图神经网络方法。同时,我们通过结合蛋白质图和图神经网络,从蛋白质序列中构建了一种结构感知图神经网络方法。
- 我们引入了自监督的预训练的药物和蛋白质的嵌入,以实现对药物和蛋白质的自我监督。为了加强蛋白质/药物关联信号,我们分别引入了药物和蛋白质的自监督预训练嵌入。我们的模型在一个统一的框架内利用这些高级信息,并通过一个基于transformer的相互作用机制产生可解释的结果。
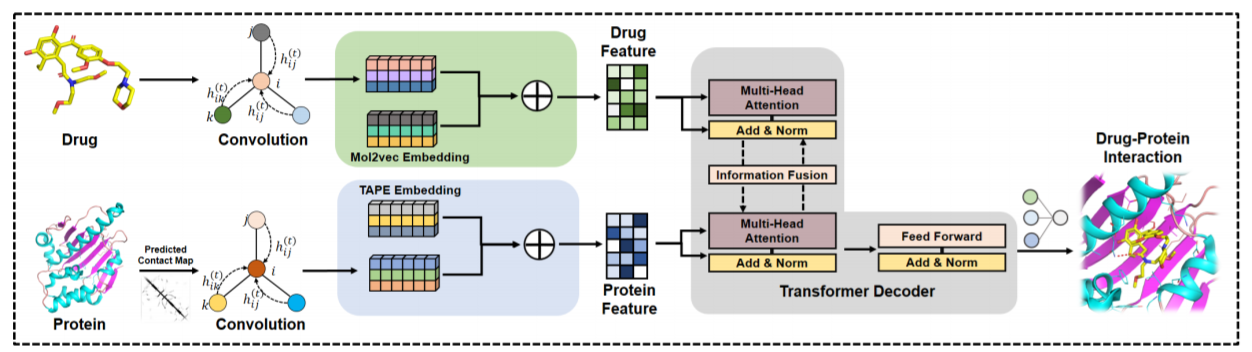
上图显示了X-DPI的架构。它首先平行处理分子和蛋白质的特征,然后将分子和蛋白质的嵌入融合在一起,通过transformer解码器进行DPI预测。
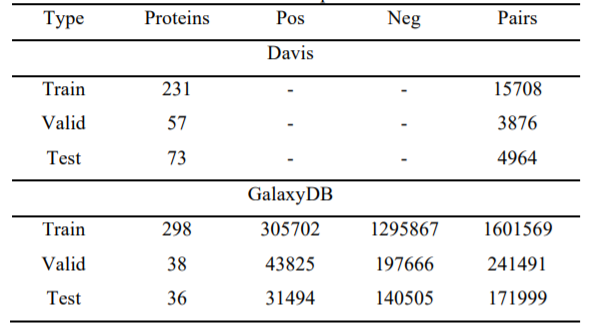
上表显示了数据集GalaxyDB的数据量和切分,分布情况。
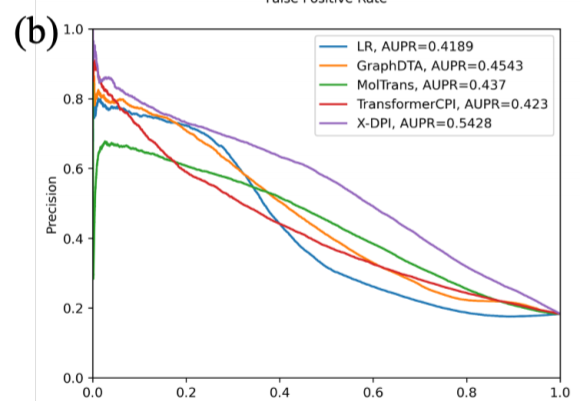
模型在GalaxyDB测试集上的表现,展示为P-R曲线。明显X-DPI效果优秀。结合在Davis数据集上的效果,可以证明该方法的效果。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢