随着遗传关联和预测研究扩展到大规模、日益多样化的生物库,描述大型队列的遗传亚结构已变得越来越重要。ADMIXTURE和STRUCTURE是广泛使用的无监督聚类算法,用于描述这种祖先的遗传结构。这些方法将单个基因组分解成分数群组分配,每个群组代表一个DNA标记频率的向量。这些分配和聚类为遗传学家提供了一个可解释的表述,以描述样本水平上的种群亚结构。然而,随着人口生物库规模的迅速扩大和每个样本基因分型(或测序)数量的增加,这种传统的方法在计算上变得难以解决。此外,使用这些传统方法需要用不同的超参数进行多次运行以正确描述群体聚类,增加了计算的负担。这可能会导致数天的计算。在这项工作中,我们提出了Neural ADMIXTURE,一个多头预训练自编码器,它遵循与ADMIXTURE相同的建模假设,提供类似的聚类,同时将计算时间减少几个数量级。此外,这个网络可以包括多个输出,提供与运行原始ADMIXTURE算法多次、不同数量的聚类相当的结果。这些模型也可以被储存起来,允许以后以线性计算时间进行聚类分配。
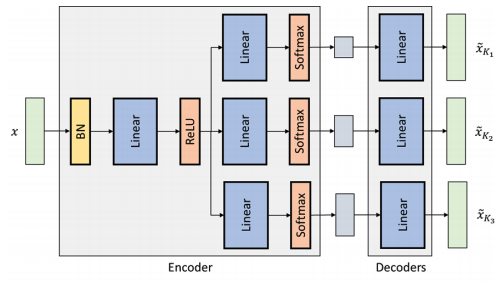
上图显示了3头的神经 ADMIXTURE架构,显示了多头结构的共享网络如何被分成H个不同的头。训练数据使用了一套全面的来自世界各地不同人群的公开的人类全基因组序列,结合了1000组基因组计划内容。非监督预训练模式允许重复使用先前优化的ADMIXTURE的结果,以加快对新数据集的推断。
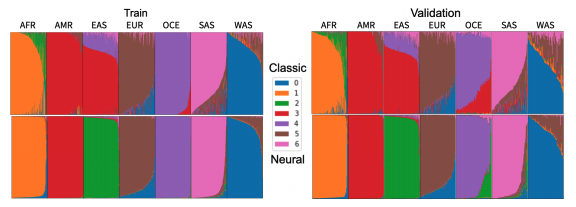
2种方法对于K=7个群组的Q估计值的可视化。每个竖条代表一个单独的样本,竖条的颜色长度代表样本的祖先被分配到该颜色聚类的比例 。上行显示的是经典的ADMIXTURE结果,下行显示的是神经 ADMIXTURE的结果显示在底部。可以看到两者总体类似,但是Neural ADMIXTURE的聚类效果更优些。
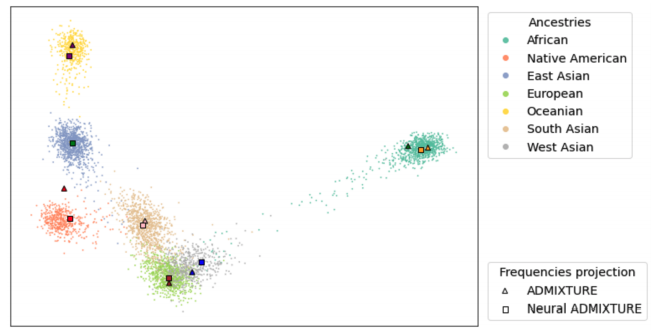
上图展示了将训练数据投射到其前两个主成分ADMIXTURE和Neural ADMIXTURE。可以看到在本土美国人和西亚人2个族群上Neural ADMIXTURE的聚类效果明显优于经典方法。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢