【论文标题】Informative RNA-base embedding for functional RNA structural alignment and clustering by deep representation learning
【作者团队】,
【通讯作者】
【发表时间】2021/08/24
【机 构】庆应义塾大学
【论文链接】https://doi.org/10.1101/2021.08.23.457433
【推荐理由】预训练RNABERT在核酸层级的表征
通过将深度学习应用于生物分子信息以获得更好的嵌入表征,可以提高下游分析的质量,如DNA序列motif检测和蛋白质功能预测。在这项研究中,作者采用了RNA碱基有效嵌入的预训练算法来获取丰富的语义表征,并将其应用于两个基本的RNA序列问题:结构比对和聚类。通过使用预训练算法RNABERT,利用大量来自不同RNA家族的RNA序列,以位置依赖的方式嵌入RNA的四个碱基,得到了一个上下文敏感的嵌入表征。训练后不仅碱基信息,而且RNA序列的二级结构和上下文信息也被嵌入每个碱基,暨此在RNA结构比对和RNA家族聚类任务中达到优于现有最先进方法的准确性。此外,通过将这种信息性碱基嵌入与简单的Needleman-Wunsch对齐算法相结合进行RNA序列对齐,我们成功地在时间复杂度为O(n2)的情况下计算出结构对齐,而不是Sankoff式算法的O(n6)时间复杂度。
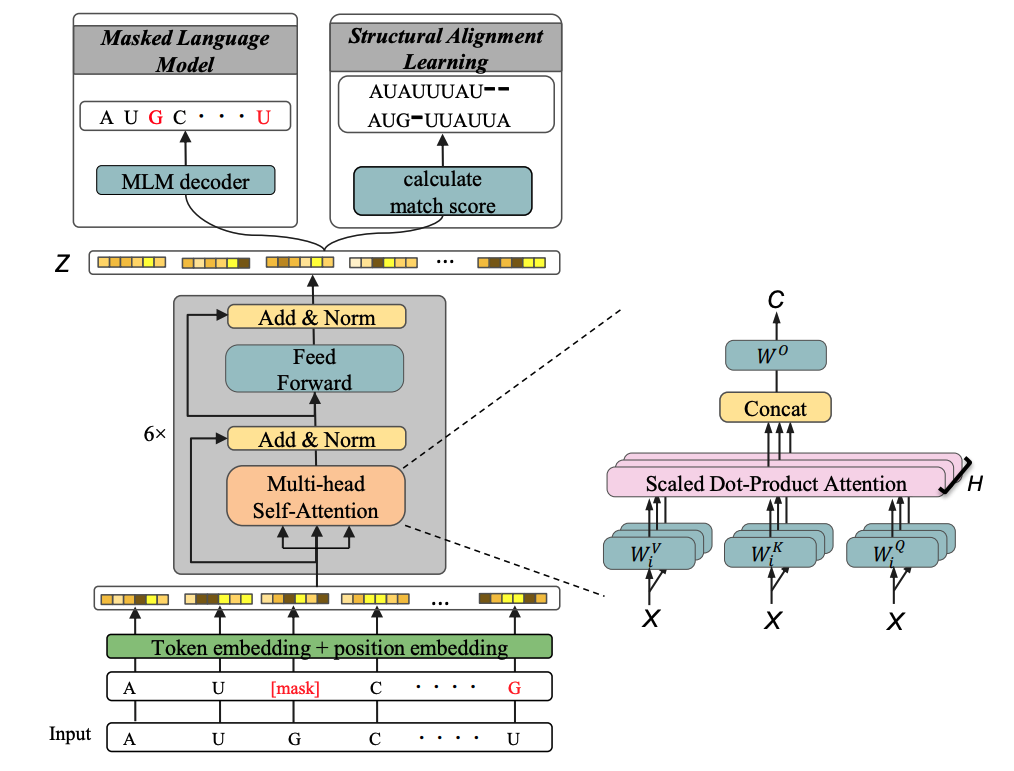
上图展示了RNABERT模型的结构。RNABERT模型由三个部分组成,标记和位置嵌入,转换层和预训练任务。标记和位置嵌入随机生成一个120维的向量,代表四个RNA碱基。变换器层组件由6个变换器层堆叠而成。每个变压器层由多头自留地组成,然后是一个前馈神经网络。变换层的权重是通过在变换层的输出之上交替进行两个不同的任务(MLM和SAL)来训练的。
SAL(structural alignment learning)任务基于Rfam数据库的比对结果,比对相似数据有相似的嵌入。

上图显示了提取的tRNA(ab)和snoRNA家族(cd)的序列motif。a图和c图是每个碱基的注意力的可视化。红色背景较深的碱基具有较高的注意力。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢