【论文标题】Hydrogen bonds meet self-attention: all you need for general-purpose protein structure embedding
【作者团队】Cheng Chen, Yuguo Zha, Daming Zhu, Kang Ning, Xuefeng Cui
【通讯作者】 Xuefeng Cui
【发表时间】2021/08/29
【机 构】山东大学,华中科大
【论文链接】https://doi.org/10.1101/2021.01.31.428935
【代码链接】https://github.com/xfcui/contactlib
【推荐理由】蛋白结构表征的基于注意力提取方式
通用的蛋白质结构嵌入可用于许多重要的蛋白质生物学任务,如蛋白质设计、药物设计和结合亲和力预测。最近的研究表明,基于注意力的编码器层更适合学习高层特征。基于这一关键观察,作者将低层次的表征学习和高层次的表征学习分开处理,并提出了一个两层次通用的蛋白质结构嵌入注意力网络,称为ContactLib-ATT。在局部嵌入层面,作者学习了一个简单而有意义的氢键表征。在全局嵌入层面上,基于注意力的编码器层被用于全局表征学习。在作者的实验中,ContactLib-ATT在SCOP40 2.07数据集上实现了82.4%的SCOP超家族分类精度(即比最先进的方法高6.7%)。此外,ContactLib-ATT被证明可以成功地模拟一个基于结构的远程同源蛋白的搜索引擎,作者的前10名候选名单至少包含一个远程同源蛋白,概率为91.9%。
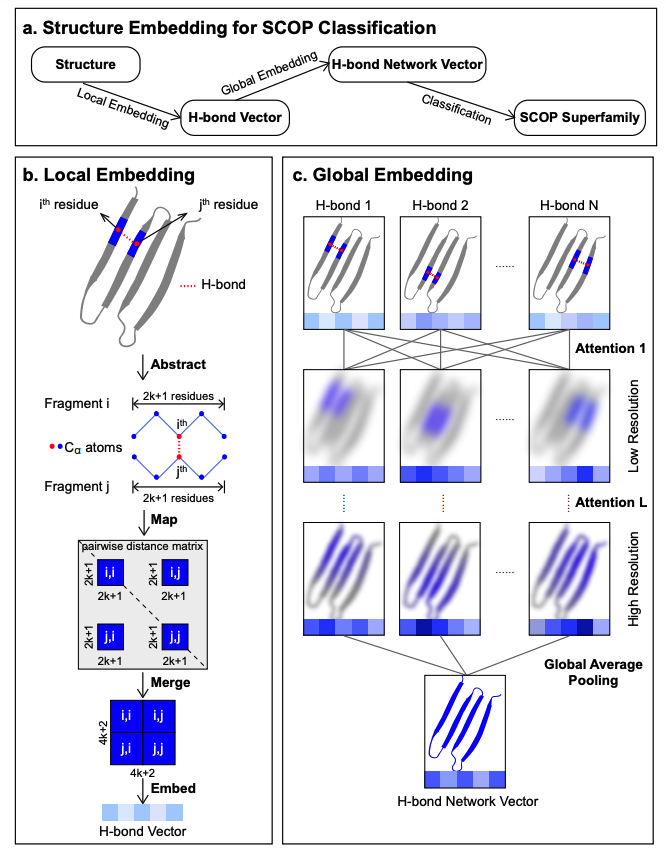
上图展示了用ContactLib-ATT进行蛋白质结构嵌入和分类:a图展示了ContactLib-ATT的流程有三个步骤;b图展示了第一个局部嵌入步骤来抽象和嵌入局部氢键(H-bond);c图展示了第二个全局嵌入步骤来嵌入一个全局H-bond网络;第三个分类步骤(在图中没有显示)作为作者通用蛋白质结构嵌入的第一个应用。
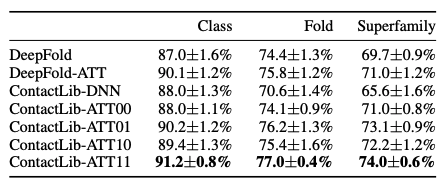
比较表二中ContactLib-DNN和ContactLib-ATT的结果,作者发现,基于注意力的全局嵌入比基于DNN的简单全局嵌入取得了更高的精度。同样地,比较DeepFold和DeepFold-ATT的结果,基于注意力的全局嵌入优于基于卷积的全局嵌入。这些观察结果与之前的研究一致,表明基于注意力的编码器层在全局特征学习方面产生了更好的结果。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢