【论文标题】Toward More General Embeddings for Protein Design: Harnessing Joint Representations of Sequence and Structure
【作者团队】Sanaa Mansoor, Minkyung Baek, Umesh Madan, Eric Horvitz
【发表时间】2021/09/01
【机 构】华盛顿大学、微软
【论文链接】https://www.biorxiv.org/content/10.1101/2021.09.01.458592v1
从序列比对中学习到的蛋白质嵌入已被用于蛋白质理解和工程的任务中。序列嵌入是通过对数以百万计的序列进行半监督训练,并使用数亿个参数的深度神经网络模型来生成的,并且它们在目标任务上的性能随着网络复杂性的增加而不断提高。我们报告了一种数据利用更高效的方法,通过对蛋白质序列和结构的联合训练,以半监督的方式编码蛋白质信息。我们表明,该方法能够对这两类信息进行编码,形成一个丰富的嵌入空间,可用于下游的预测任务。我们同时表明,将丰富的结构信息纳入所考虑的背景中,通过预测单次突变的影响,提高了模型的性能。作者将准确率的提高归功于利用丰富的表征来确定将受到突变影响的顺序和空间上接近的残基,使用实验验证的或预测的结构。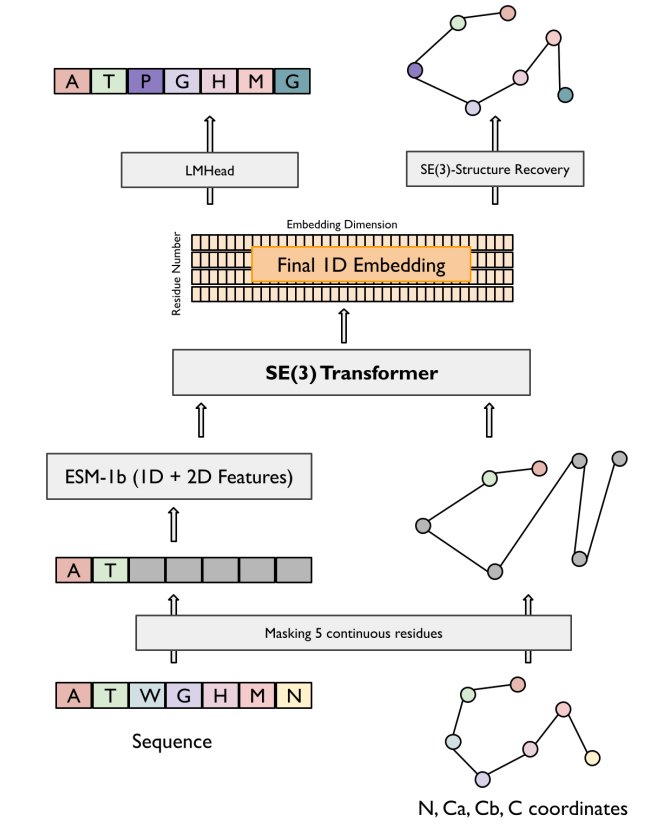
上图显示了如何构建和利用联合使用序列和结构信息的蛋白质嵌入的模型架构。首先对相同的序列和结构区域进行屏蔽,通过预训练的ESM-1b模型对被掩蔽的序列进行处理,产生一维和二维特征,屏蔽后的结构和序列表示被作为输入传给SE(3)Transformer,输出一个128维的嵌入空间,嵌入空间被用来预测被屏蔽的序列和结构区域。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢