【论文标题】GPT-3 Models are Poor Few-Shot Learners in the Biomedical Domain
【作者团队】Milad Moradi, Kathrin Blagec, Florian Haberl, Matthias Samwald
【发表时间】2021/09/06
【机 构】维也纳大学
【论文链接】https://arxiv.org/abs/2109.02555v1
深度神经语言模型在自然语言处理的许多任务中都取得了新的突破。最近的工作表明,在大量文本上进行了预训练的深度Transformer语言模型可以实现与最先进的模型相媲美的高水平的特定任务小样本性能。然而,在生物医学领域,这些大型语言模型的小样本迁移学习的能力还没有被探索。本文研究了两个强大的Transformer语言模型,即GPT-3和BioBERT,在各种生物医学NLP任务的小样本配置中的表现。实验结果表明,在很大程度上,这两个模型的表现都不如在完整训练数据上进行微调的语言模型。尽管GPT-3已经在开放领域NLP任务上的小样本知识迁移中取得了接近最先进的结果,但它不能像BioBERT那样有效,而BioBERT比GPT-3小几个数量级。关于BioBERT已经在大型生物医学文本语料库上进行了预训练,本文的研究表明,语言模型可能在很大程度上受益于特定任务的小样本学习的域内预训练。然而,领域内的预训练似乎还不够,在生物医学NLP领域需要新的预训练和小样本学习策略。
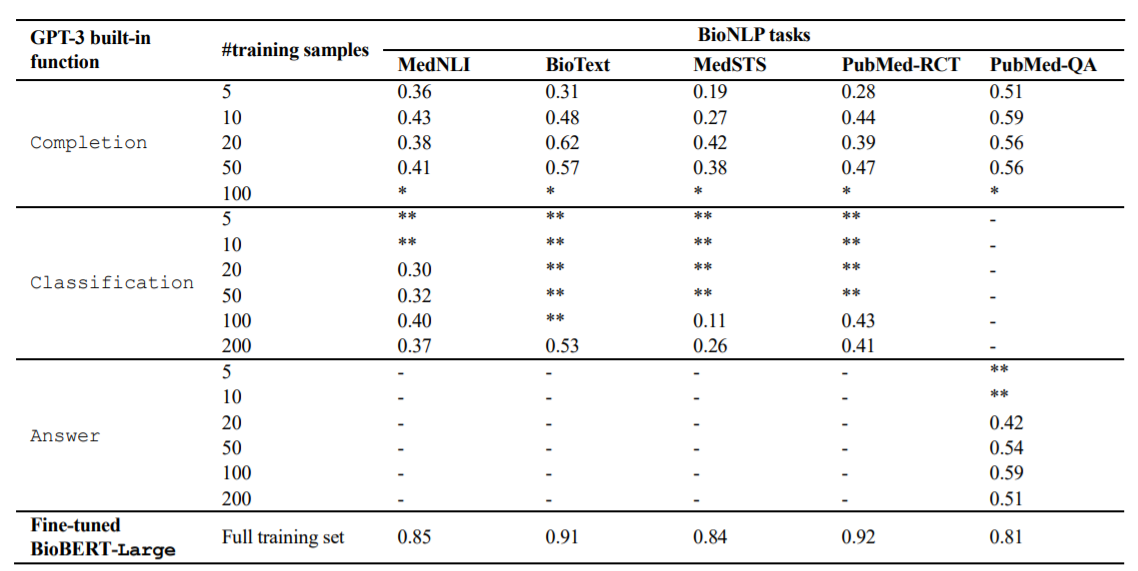
上图展示了GPT-3在BioNLP任务中使用三种不同的内置函数在小样本情况下的表现。在第一轮实验中,本文使用了OpenAI API的通用内置函数Completion,它可以用于各种任务。它接收一个prompt,其中包含任务的文字描述和一些训练样本,这样每个样本都有一个上下文和一个期望的完成度,以指示模型的输出应该是什么样子。在第二轮实验中,本文使用了另外两个为特定任务设计的内置函数。本文使用Classification功能在MedNLI、BioText、MedSTS和PubMed-CT任务上运行模型,这个函数接收一个查询和一组标记的样本,通过样本搜索来找到最相关的样本,并对其进行预测。为了执行PubMedQA任务,本文使用了Answer函数,它根据所提供的上下文和一组训练样本回答输入的问题。
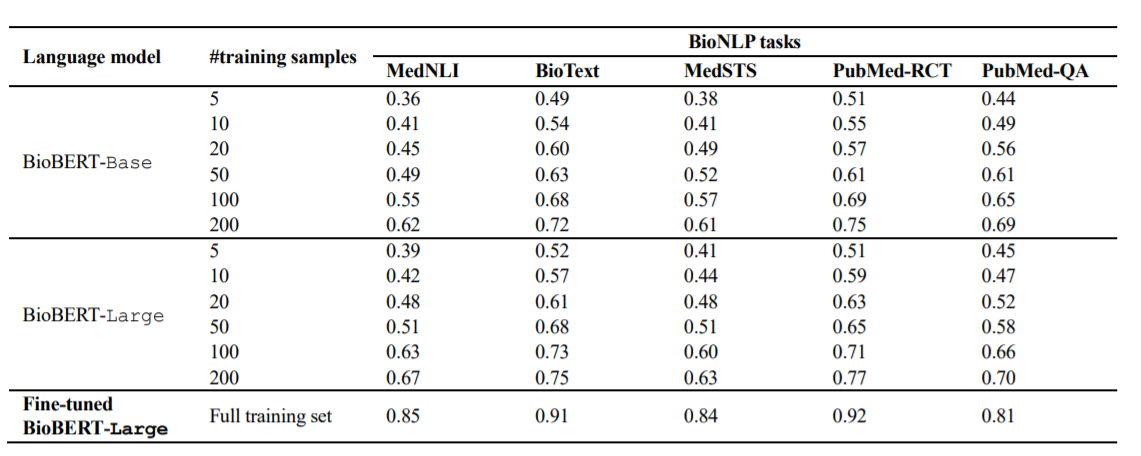
BioBERT在使用基础语言模型和大型语言模型的情况下,在小样本的配置中对BioNLP任务的表现。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢